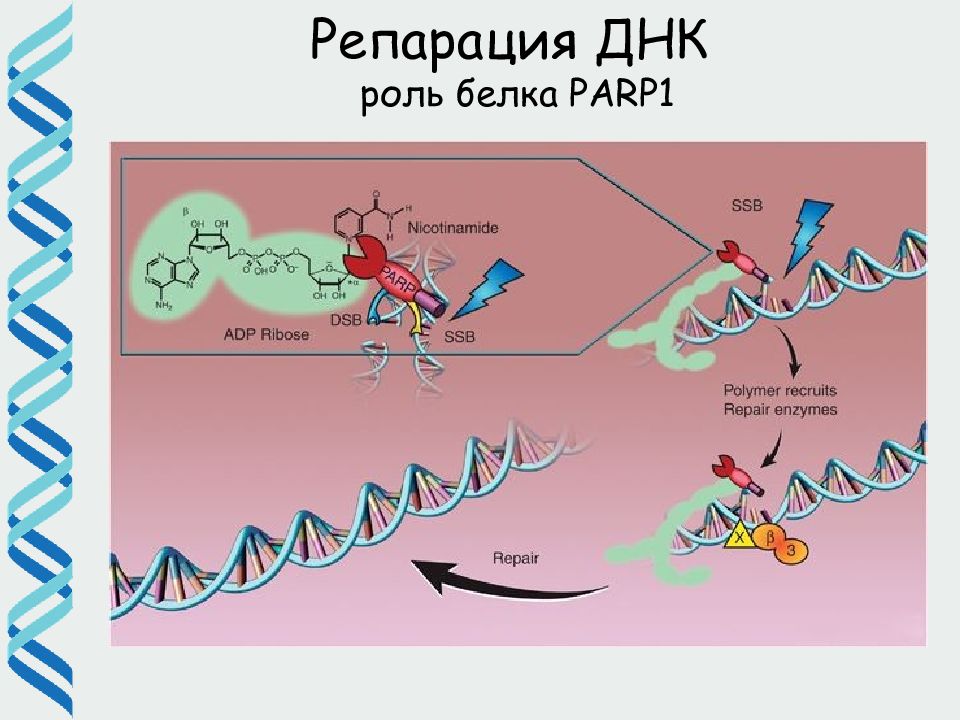
Слайд 2
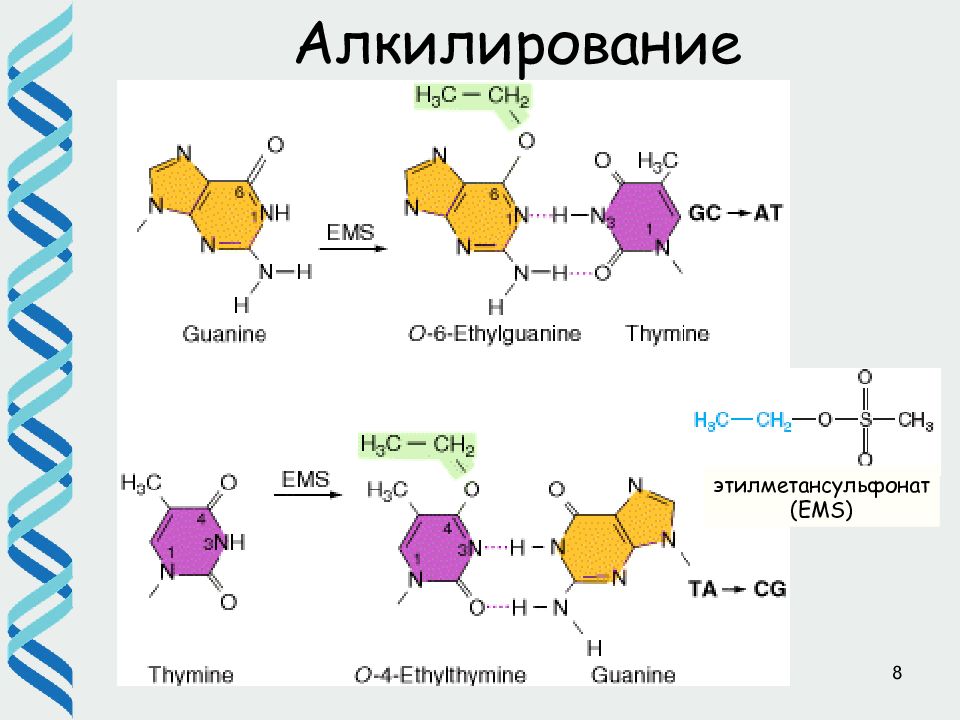
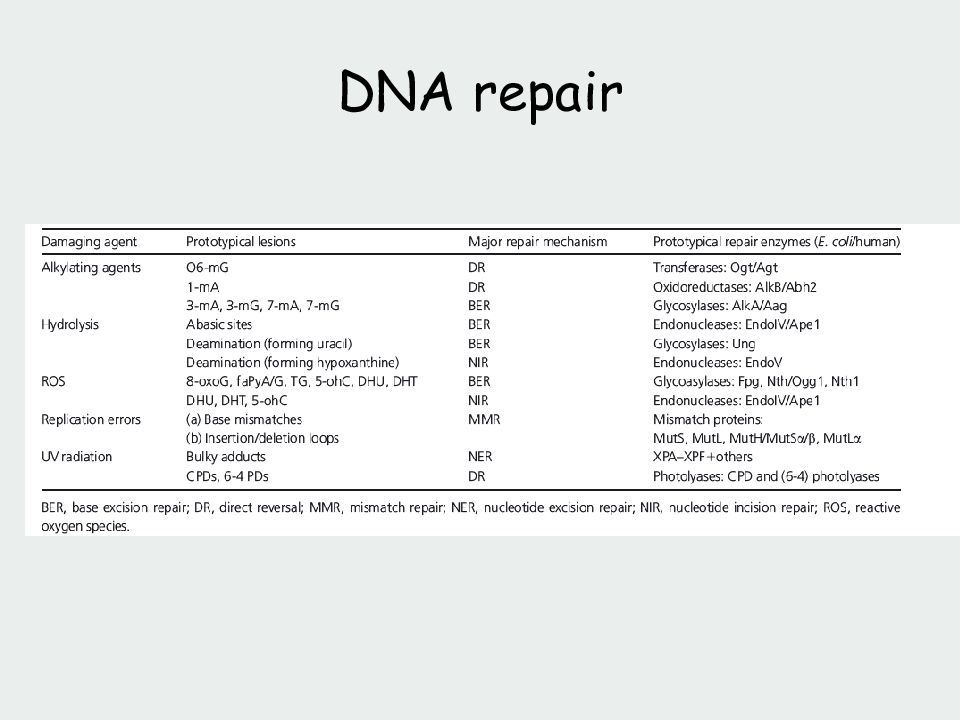
2 Причины появления повреждений в ДНК • Ошибки репликации • Повреждения ДНК эндогенными агентами гидролиз (депуринизация, дезаминирование) • Повреждения ДНК экзогенными агентами облучение повреждение химическими агентами (например, алкилирование) • Репликация «через повреждения» с использованием полимераз, отличающихся низкой точностью копирования Повреждение ДНК – любое изменение ДНК, которое вызывает отклонение от двуцепочечной структуры
Слайд 3
3 Типы повреждений ДНК На уровне одного нуклеотида • Отсутствие основания • Некомплементарное основание • Основание с нарушенной структурой Структурные • Одноцепочечные разрывы • Неспецифические связи между цепями • Двухцепочечные разрывы
Слайд 5
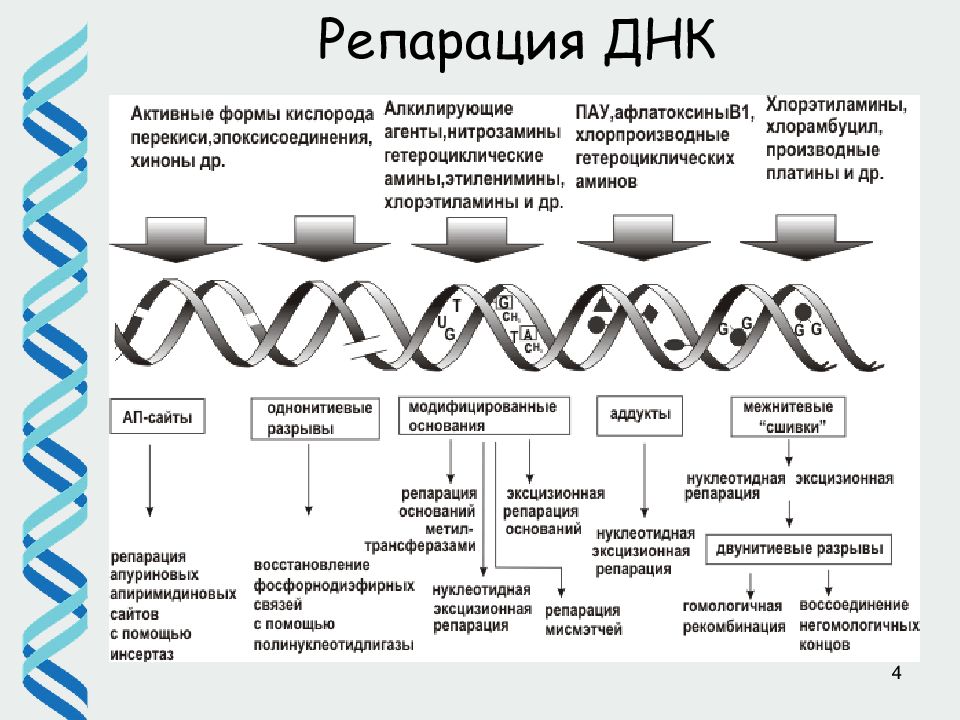
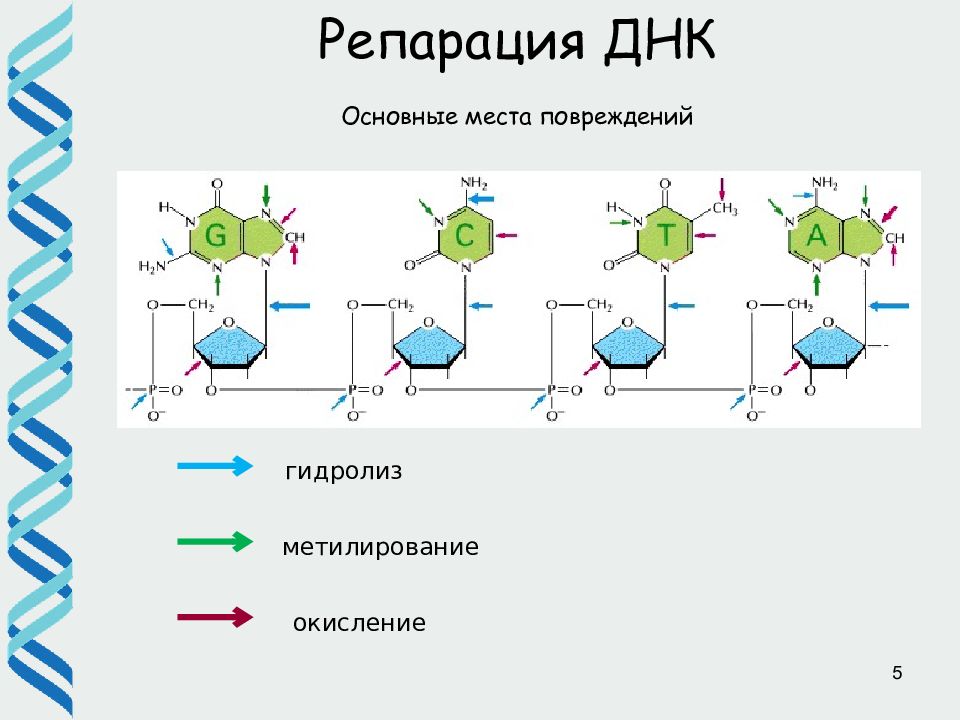
5 Репарация ДНК гидролиз метилирование окисление Основные места повреждений
Слайд 6
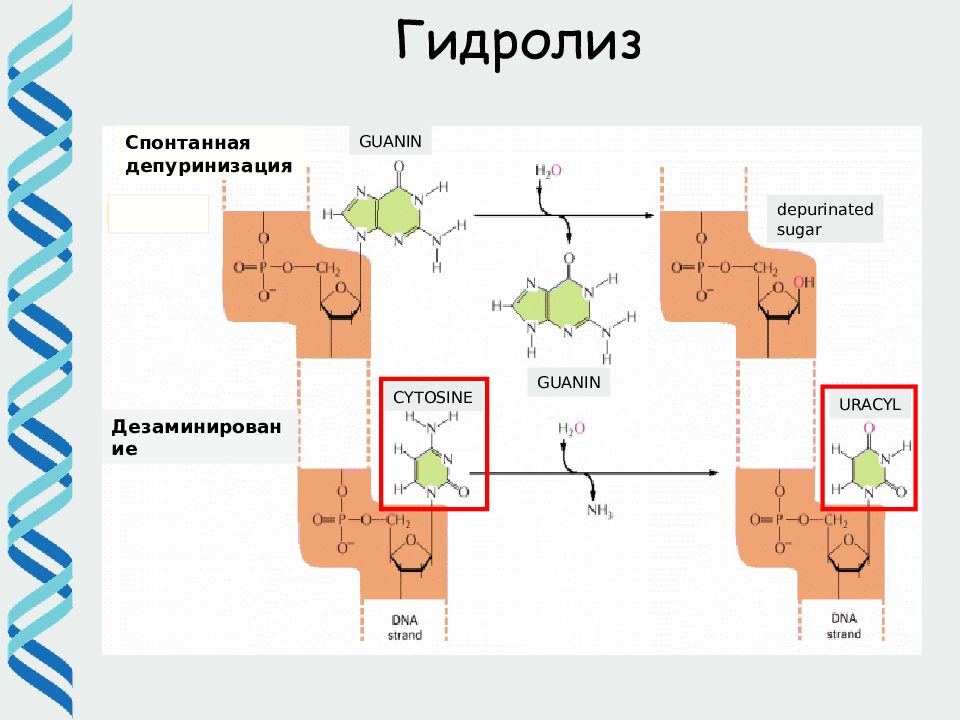
Спонтанная депуринизация Дезаминирование GUANIN GUANIN CYTOSINE URACYL depurinated sugar Гидролиз
Слайд 7
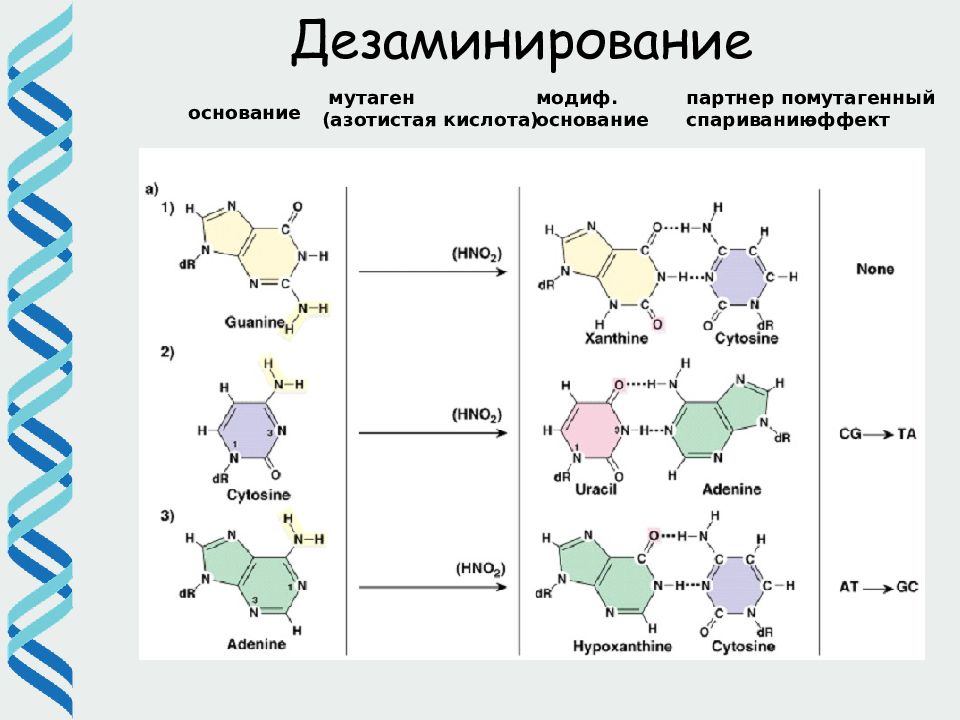
Дезаминирование основание мутаген (азотистая кислота) модиф. основание партнер по спариванию мутагенный эффект
Слайд 9
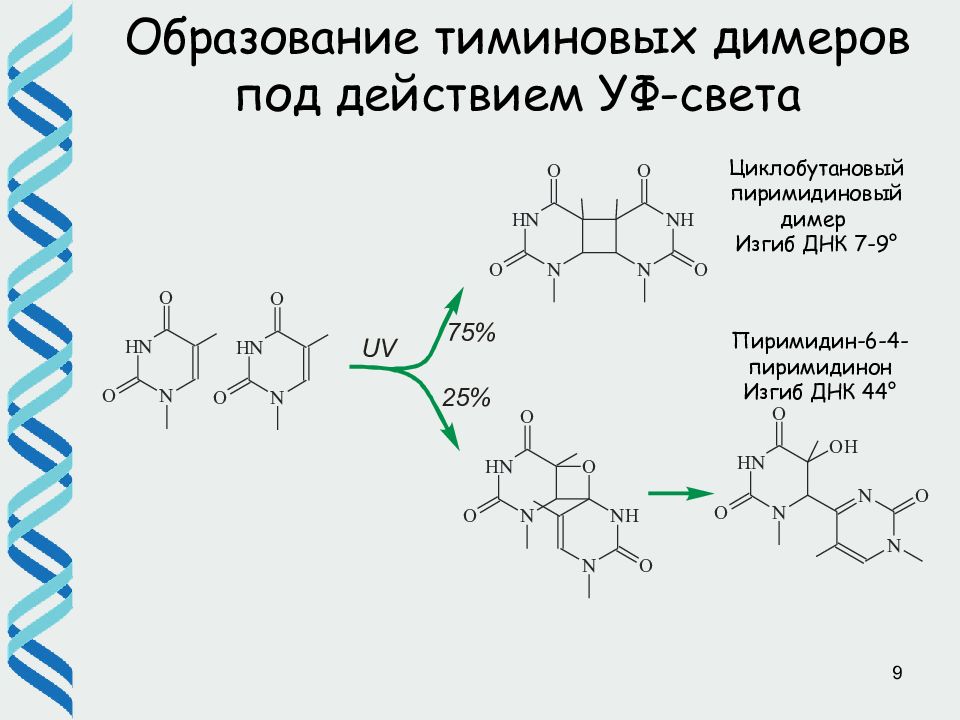
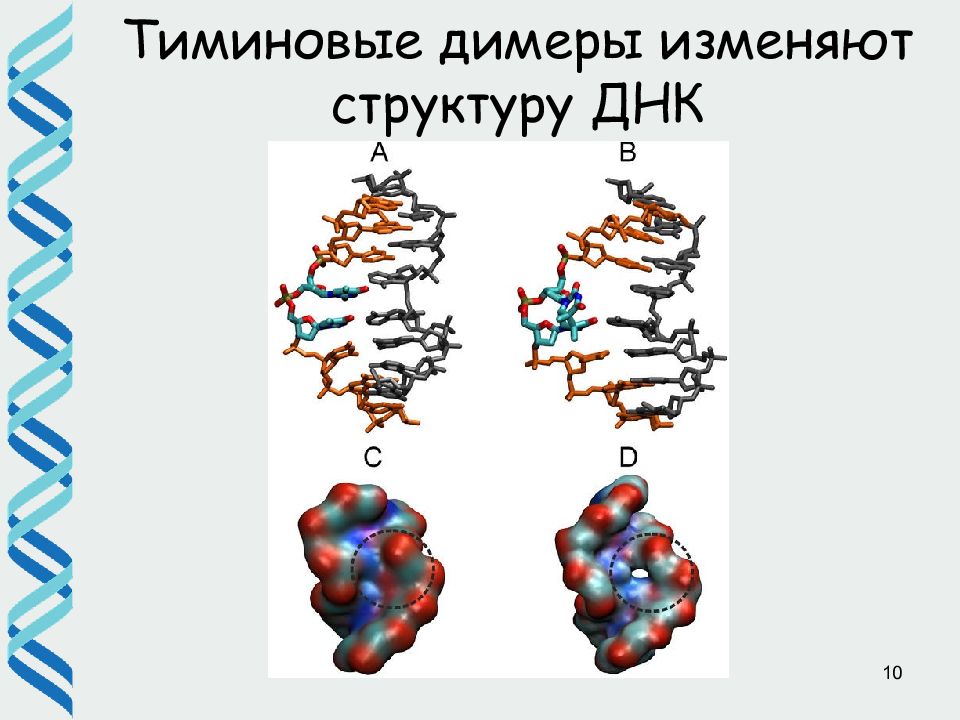
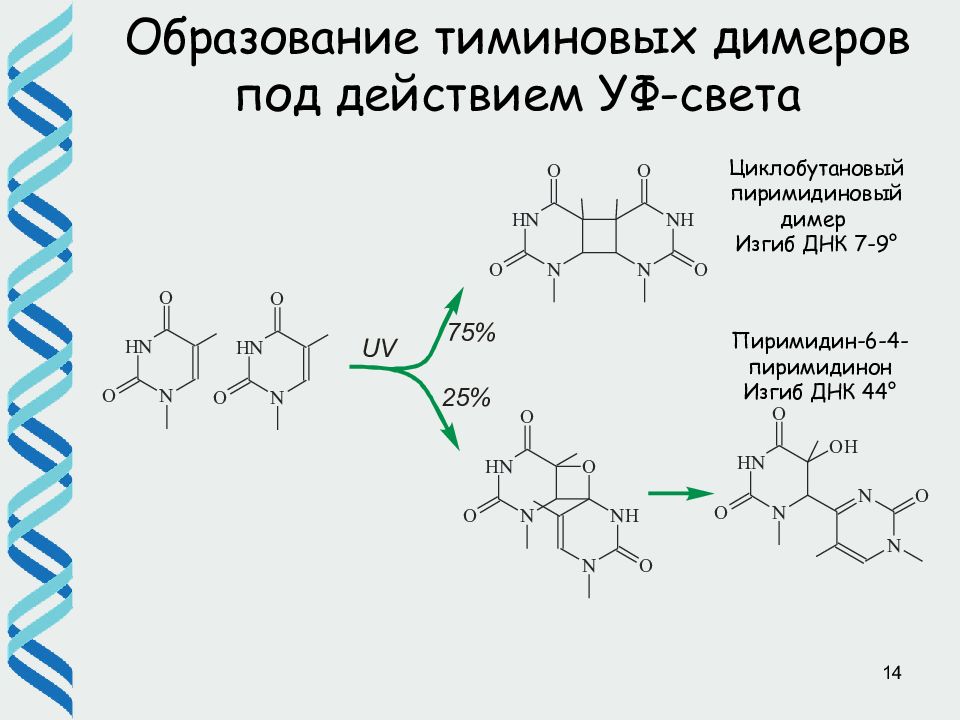
9 Образование тиминовых димеров под действием УФ-света Циклобутановый пиримидиновый димер Изгиб ДНК 7-9 ° Пиримидин-6-4-пиримидинон Изгиб ДНК 44 °
Слайд 11
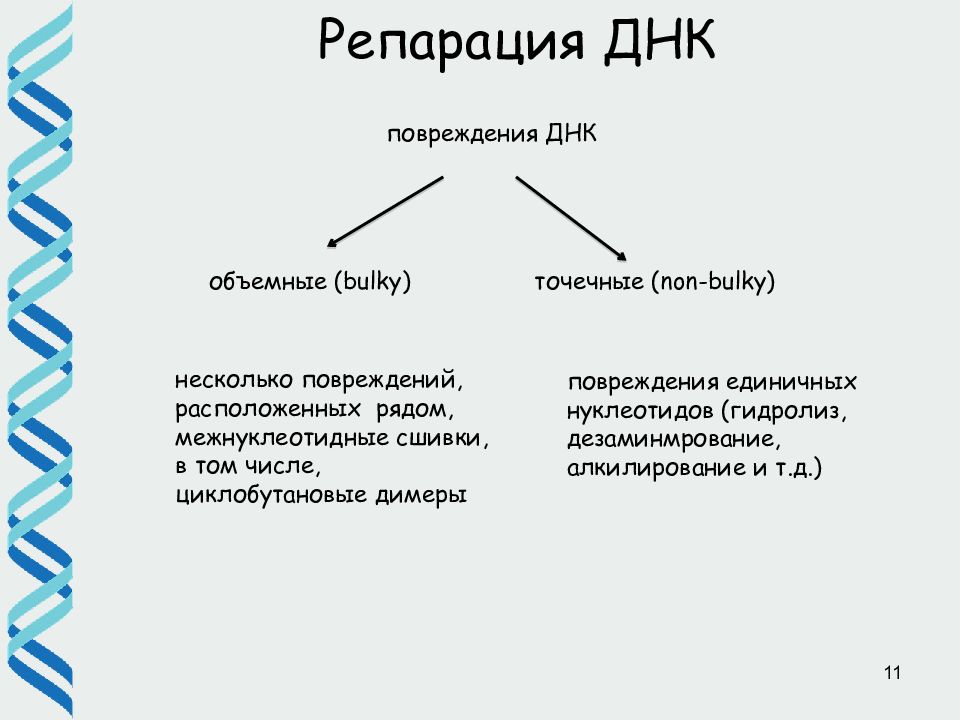
11 Репарация ДНК несколько повреждений, расположенных рядом, межнуклеотидные сшивки, в том числе, циклобутановые димеры повреждения единичных нуклеотидов (гидролиз, дезаминмрование, алкилирование и т.д.) повреждения ДНК объемные ( bulky) точечные ( non-bulky)
Слайд 12
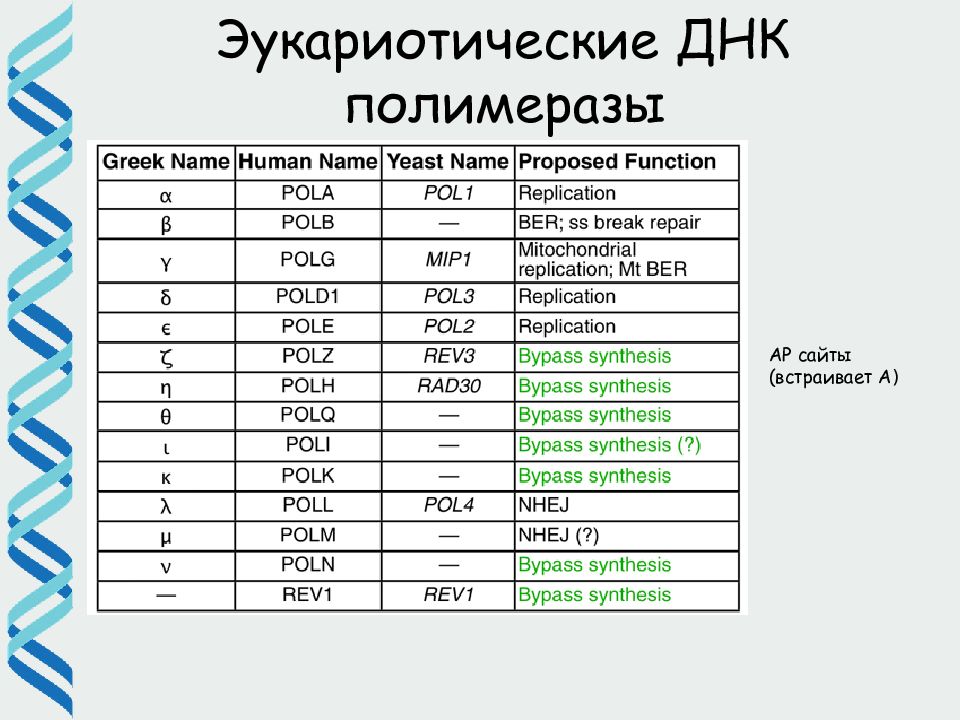
12 Репарация ДНК Стратегии коррекции повреждений Ошибки репликации : исправление ошибок ДНК полимеразой ( 3 ’ -5 ’ экзонуклеаза), репарация неспареных оснований ( mismatch repair ) Повреждение ДНК эндогенными и экзогенными агентами : Прямое удаление повреждений Вырезание (эксцизия) оснований ( base excision repair ) Вырезание (эксцизия) нуклеотидов ( nucleotide excision repair ) Рекомбинация [ Черезблоковый синтез особыми полимеразами (не удаляет ошибок но позволяет продолжить репликацию) ]
Слайд 13
13 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
Слайд 14
14 Образование тиминовых димеров под действием УФ-света Циклобутановый пиримидиновый димер Изгиб ДНК 7-9 ° Пиримидин-6-4-пиримидинон Изгиб ДНК 44 °
Слайд 15
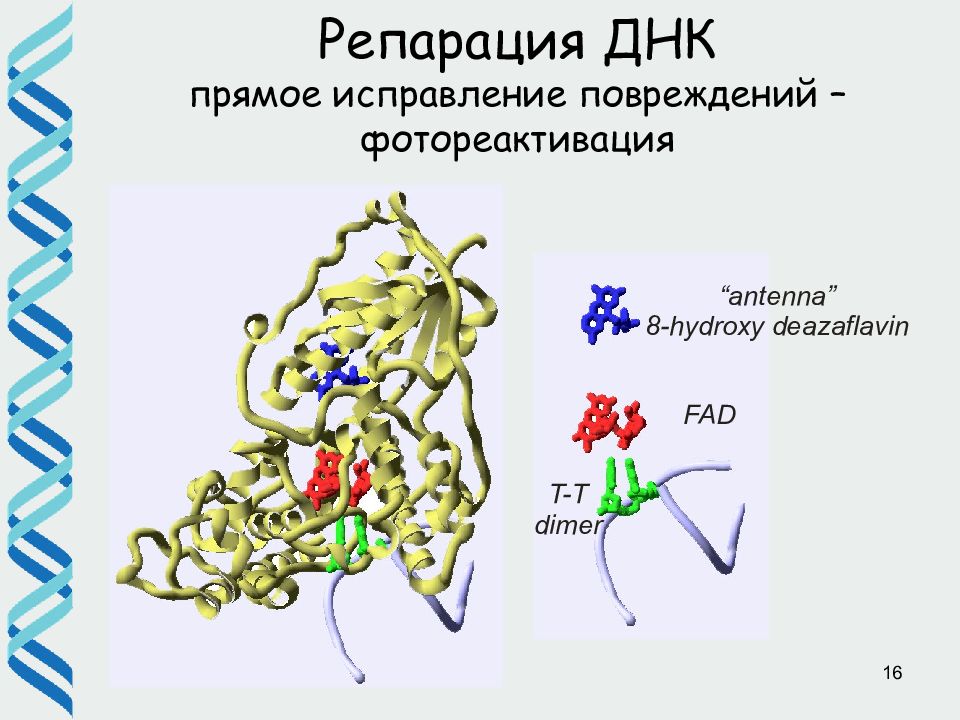
15 Репарация ДНК прямое исправление повреждений – фотореактивация ДНК-фотолиазы, мономерные флавин-зависимый ферменты Кофакторы : FADH - и 5,10-метенилтетрагидрофолат (5,10-MTHF) Фотолиаза с вязывается в темноте с димерами ТТ На свету кофактор (5,10-MTHF) абсорбирует фотон и передает энергию возбуждения на FADH - Возбужденный FADH - отдает электрон пиримидиновому димеру, устраняя повреждение Фотолиаза освобождает ДНК В клетках млекопитающих не найдены!
Слайд 18
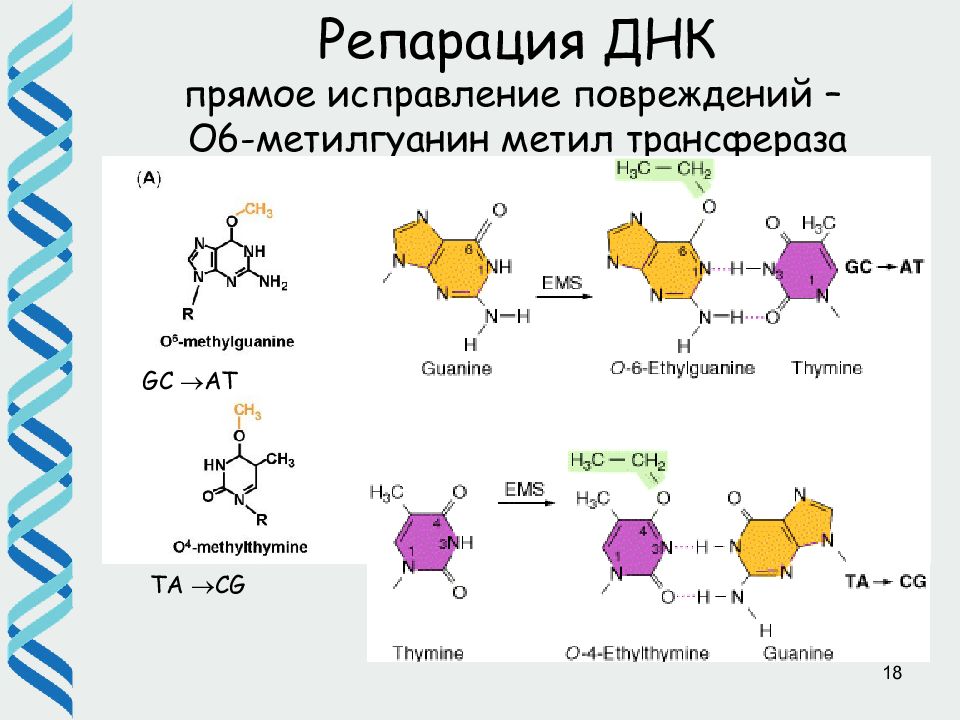
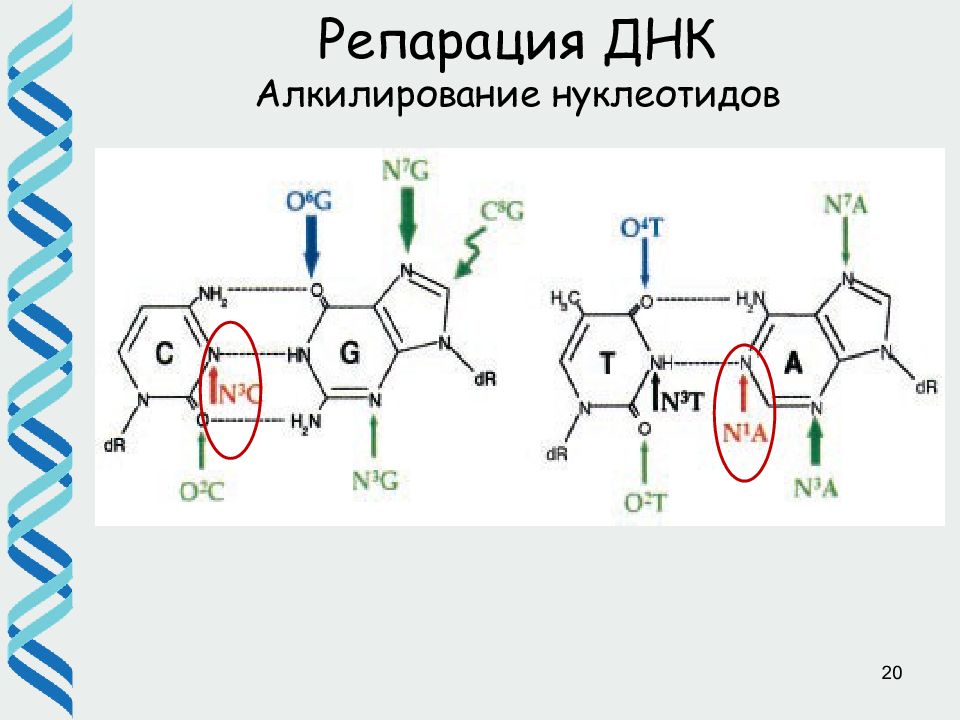
18 S N 2 mechanism TA CG GC AT Репарация ДНК прямое исправление повреждений – О6-метилгуанин метил трансфераза Ada (E.coli)
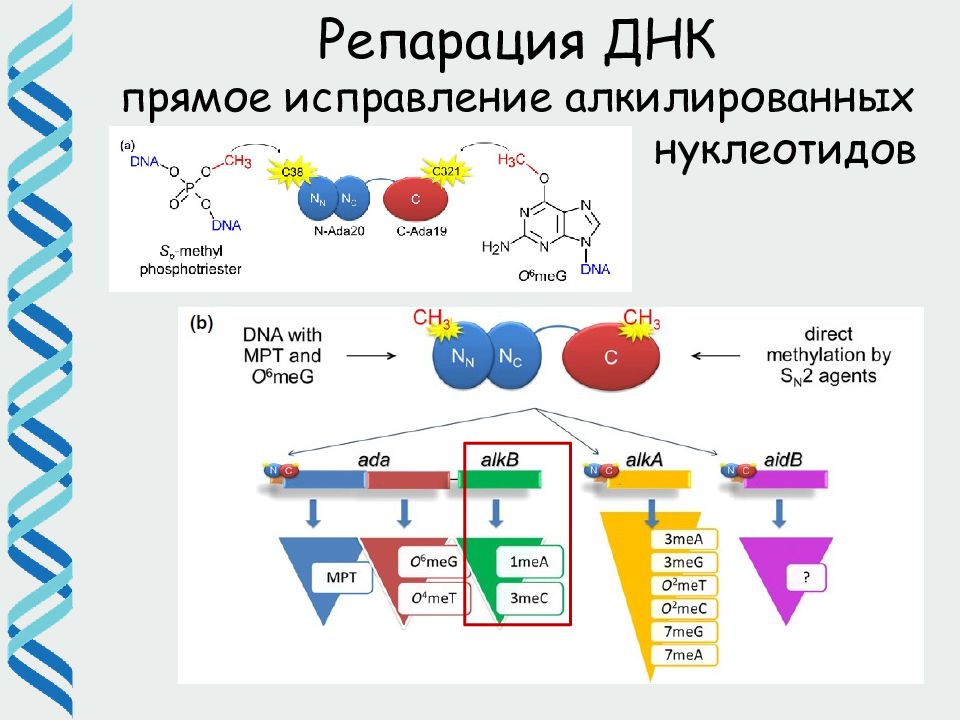
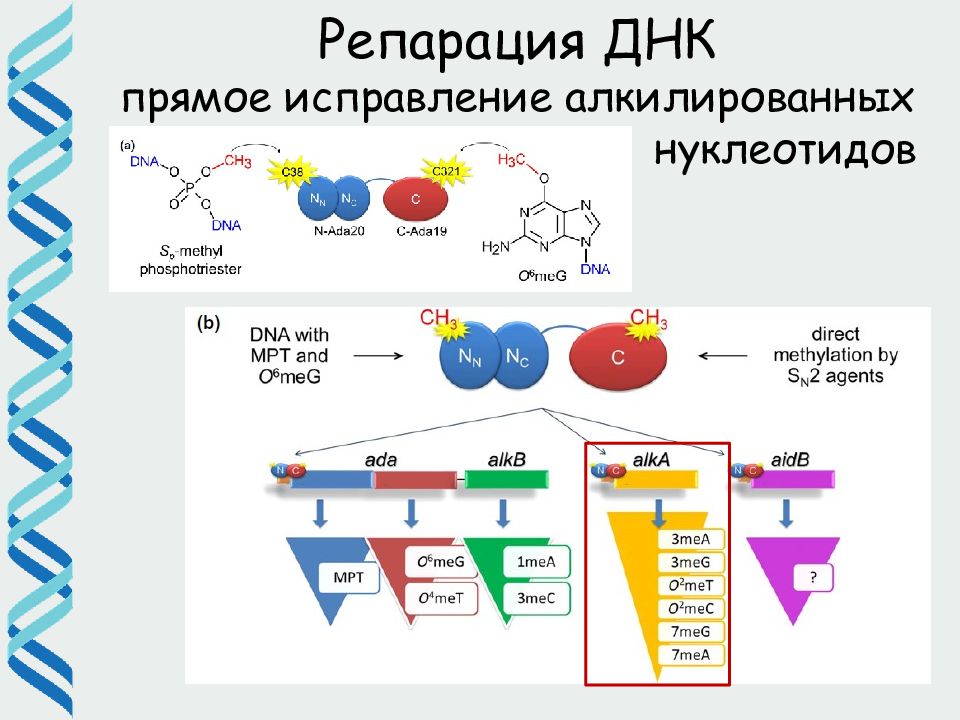
Слайд 21
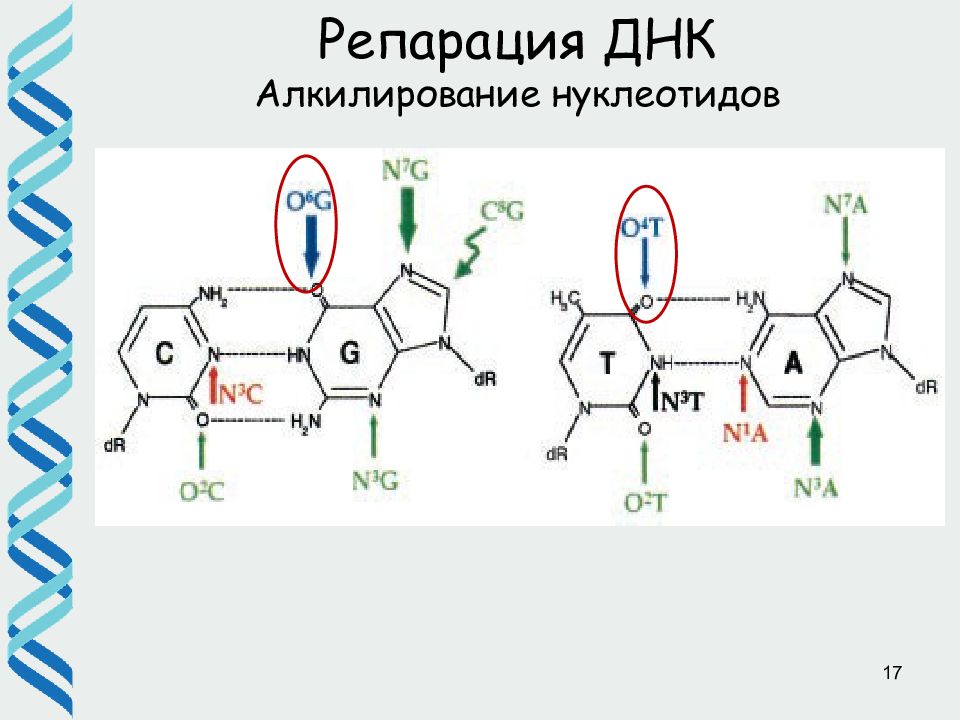
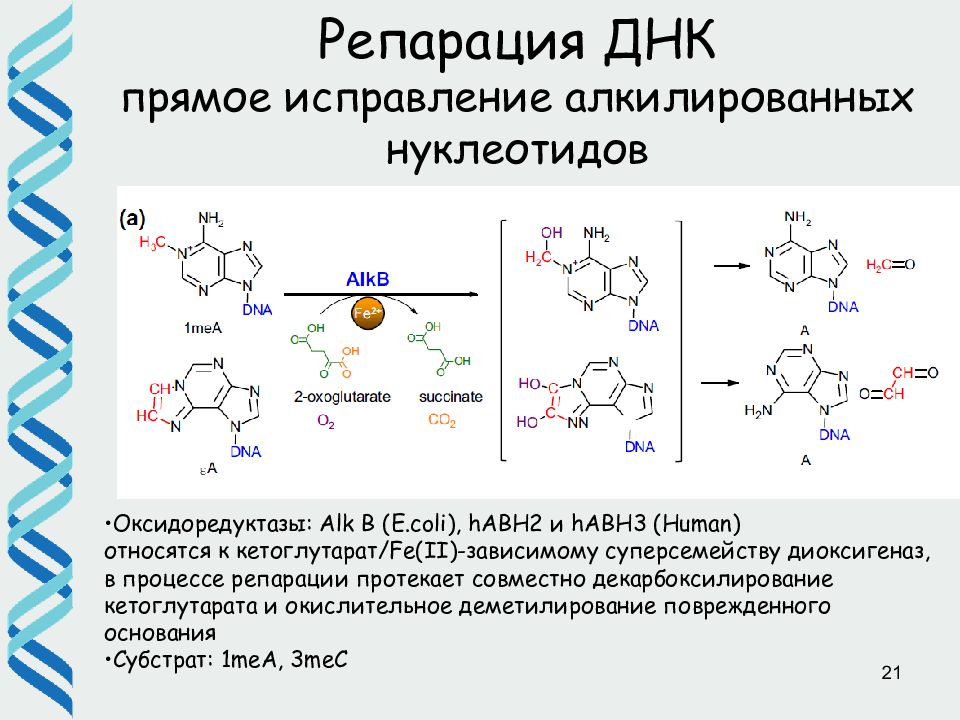
21 Репарация ДНК прямое исправление алкилированных нуклеотидов •Оксидоредуктазы: Alk B (E.coli), hABH2 и hABH3 ( Human) относятся к кетоглутарат/Fe(II)-зависимому суперсемейству диоксигеназ, в процессе репарации протекает совместно декарбоксилирование кетоглутарата и окислительное деметилирование поврежденного основания •Субстрат: 1 meA, 3meC
Слайд 25
25 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
Слайд 26
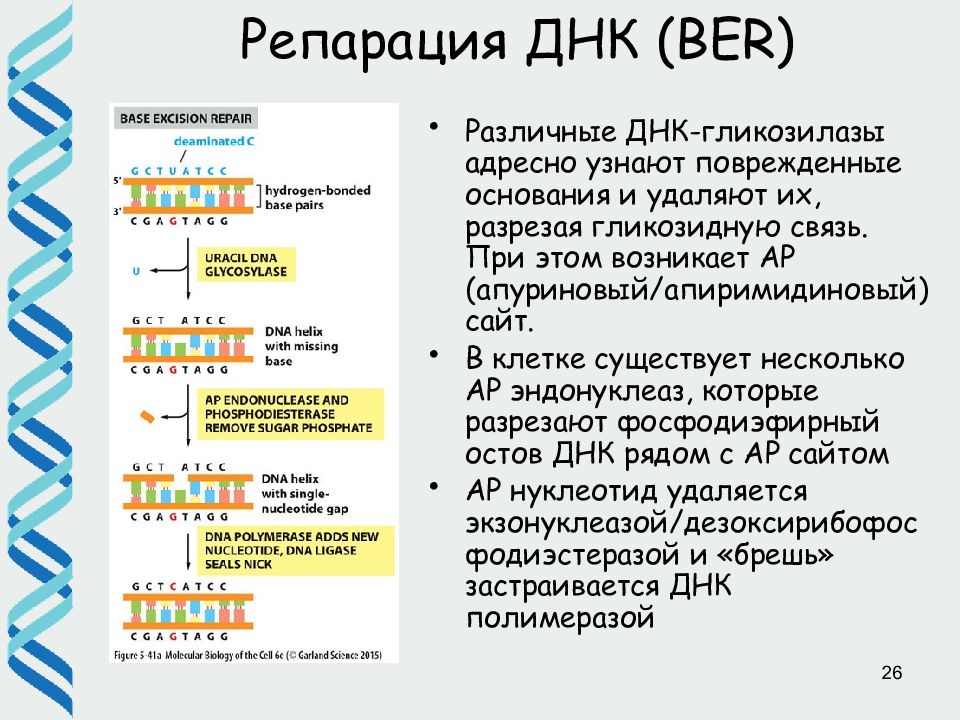
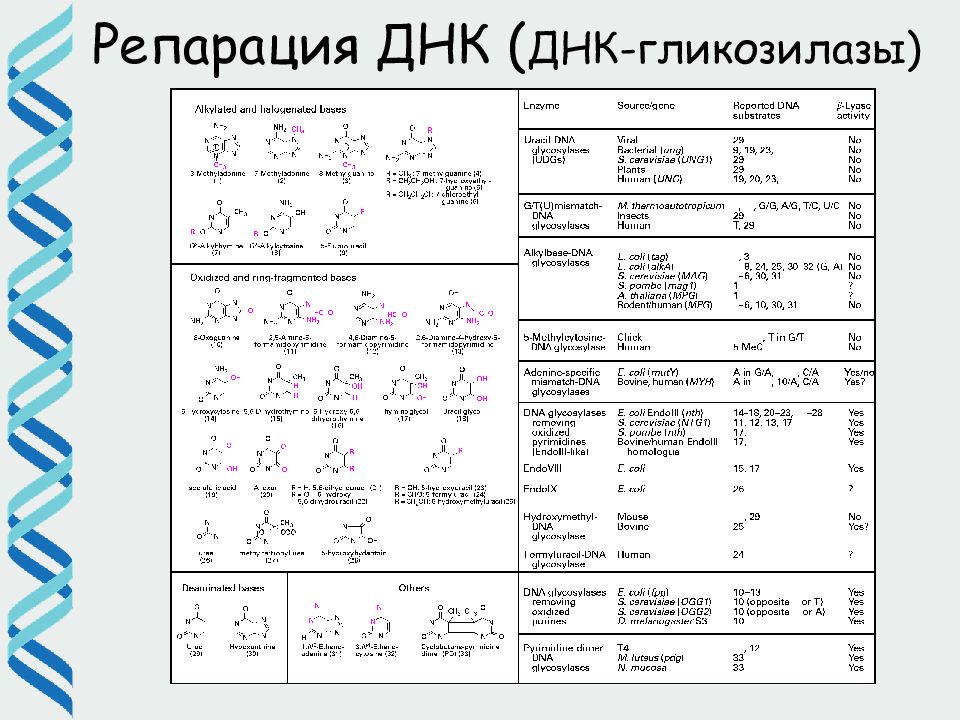
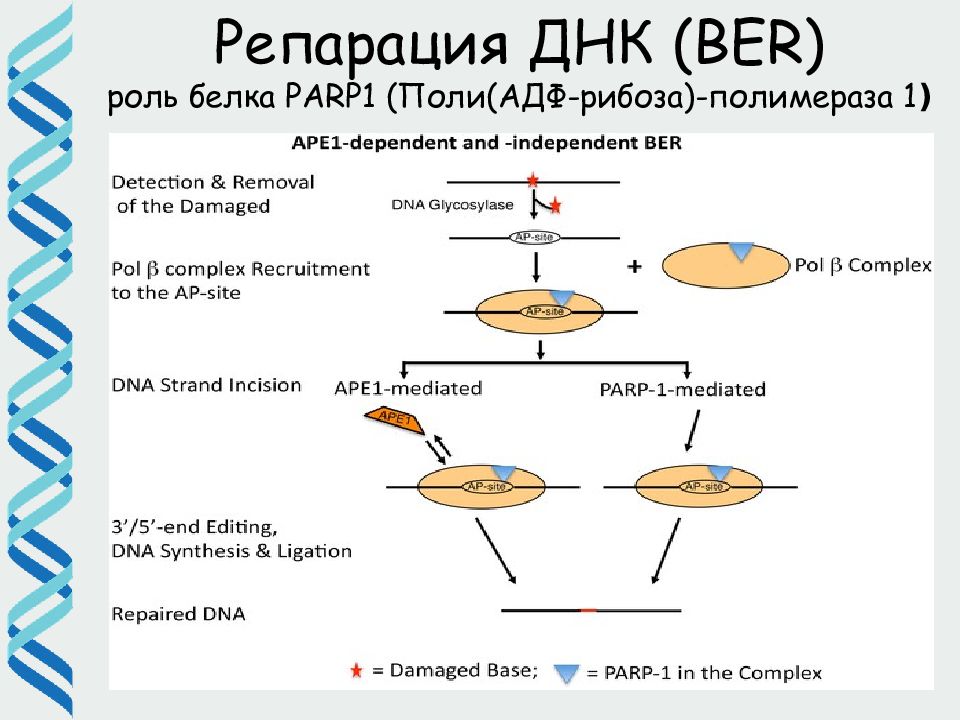
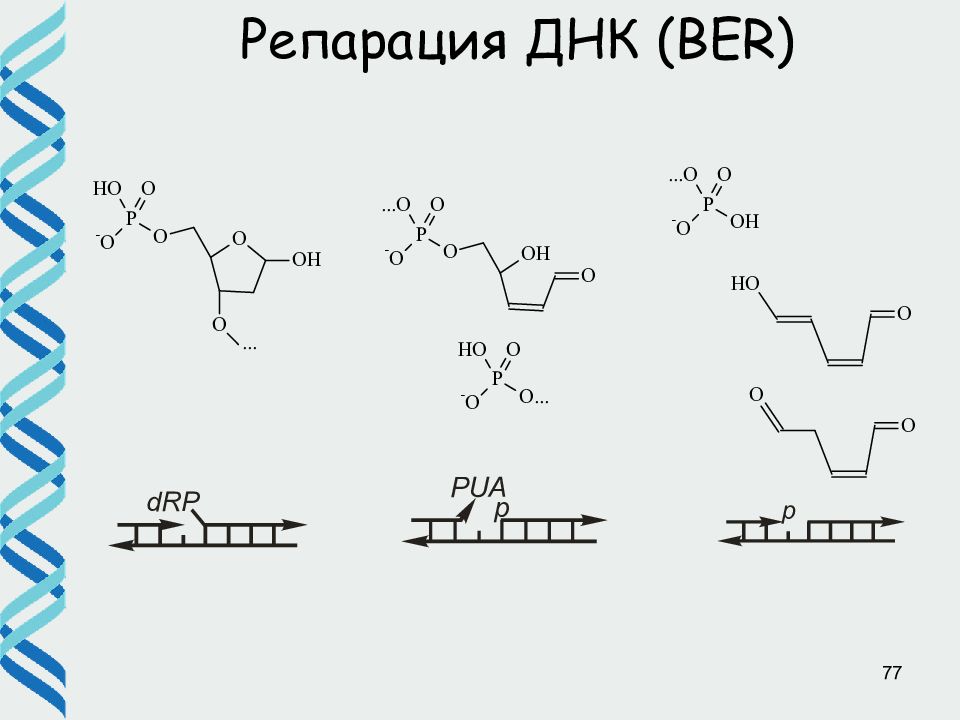
26 Репарация ДНК ( BER) Различные ДНК-гликозилазы адресно узнают поврежденные основания и удаляют их, разрезая гликозидную связь. При этом возникает AP (апуриновый/апиримидиновый) сайт. В клетке существует несколько АР эндонуклеаз, которые разрезают фосфодиэфирный остов ДНК рядом с AP сайтом AP нуклеотид удаляется экзонуклеазой / дезоксирибофосфодиэстеразой и «брешь» застраивается ДНК полимеразой
Слайд 27
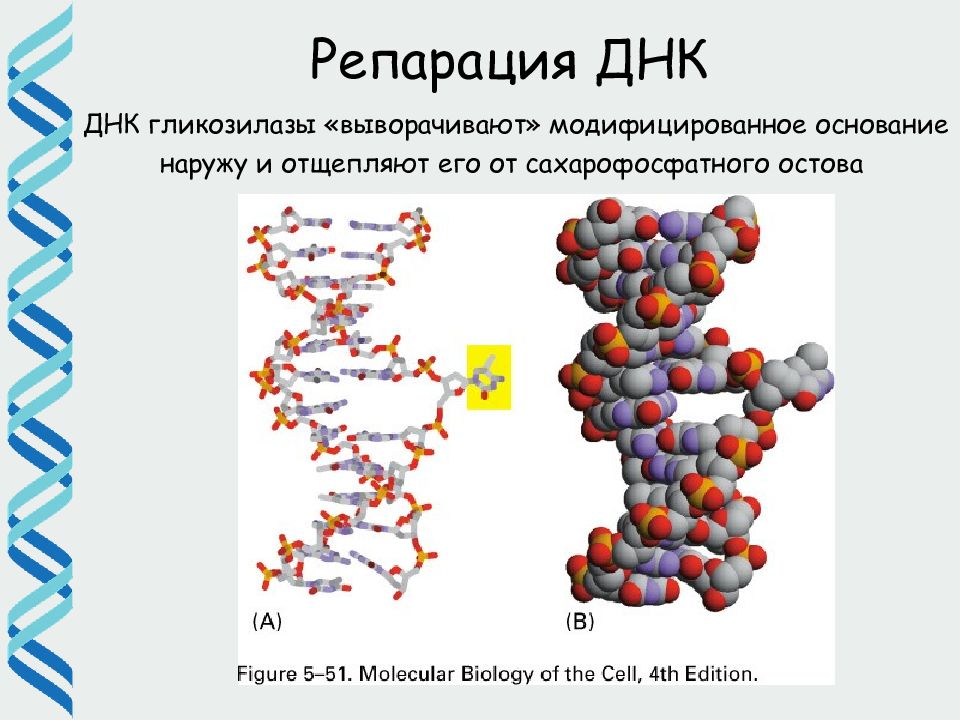
ДНК гликозилазы «выворачивают» модифицированное основание наружу и отщепляют его от сахарофосфатного остова Репарация ДНК
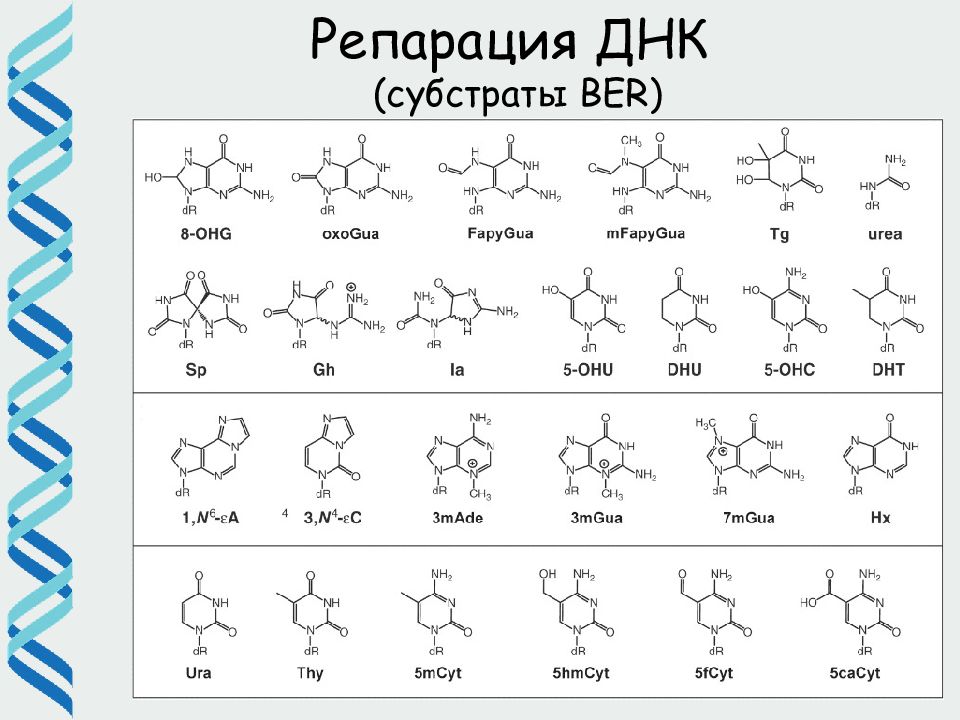
Слайд 29: Основные типы повреждений, которые удаляются посредством BER (большая часть не блокирует репликацию)
Окисленные основания, в том числе 8-o кси -G, который спаривается с А, вызывая GC --> TA трансверсии Дезоксиурацил Различные продукты алкилирования оснований (например, 3-meA ) Спонтанно возникающие апуриновые сайты
Слайд 31
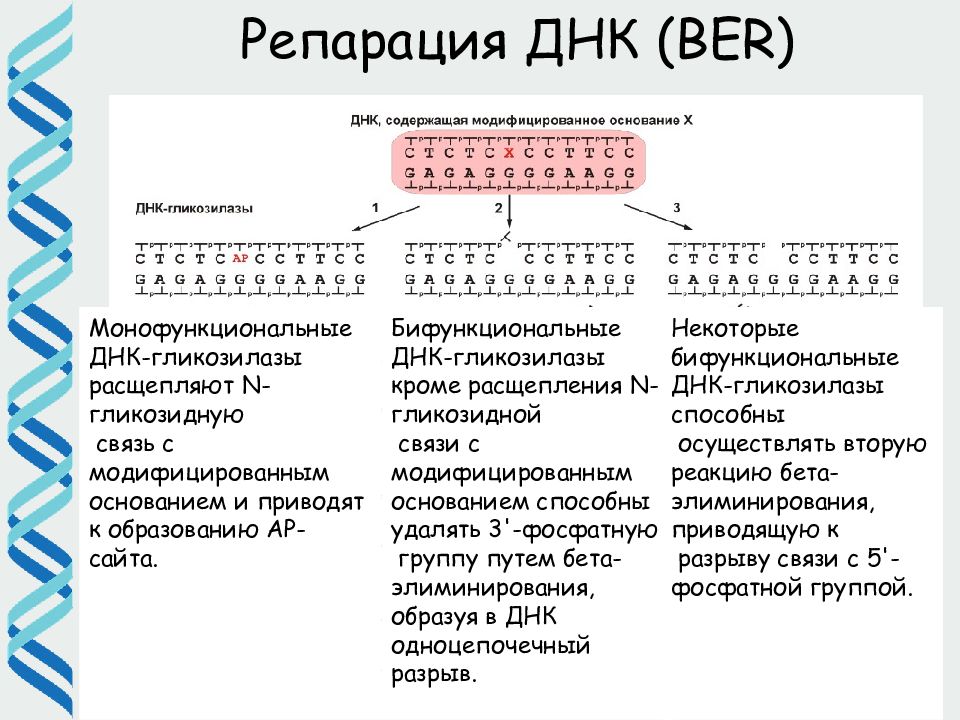
31 Репарация ДНК ( BER) Монофункциональные ДНК-гликозилазы расщепляют N-гликозидную связь с модифицированным основанием и приводят к образованию АР-сайта. Бифункциональные ДНК-гликозилазы кроме расщепления N -гликозидной связи с модифицированным основанием способны удалять 3'-фосфатную группу путем бета-элиминирования, образуя в ДНК одноцепочечный разрыв. Некоторые бифункциональные ДНК-гликозилазы способны осуществлять вторую реакцию бета-элиминирования, приводящую к разрыву связи с 5'-фосфатной группой.
Слайд 32
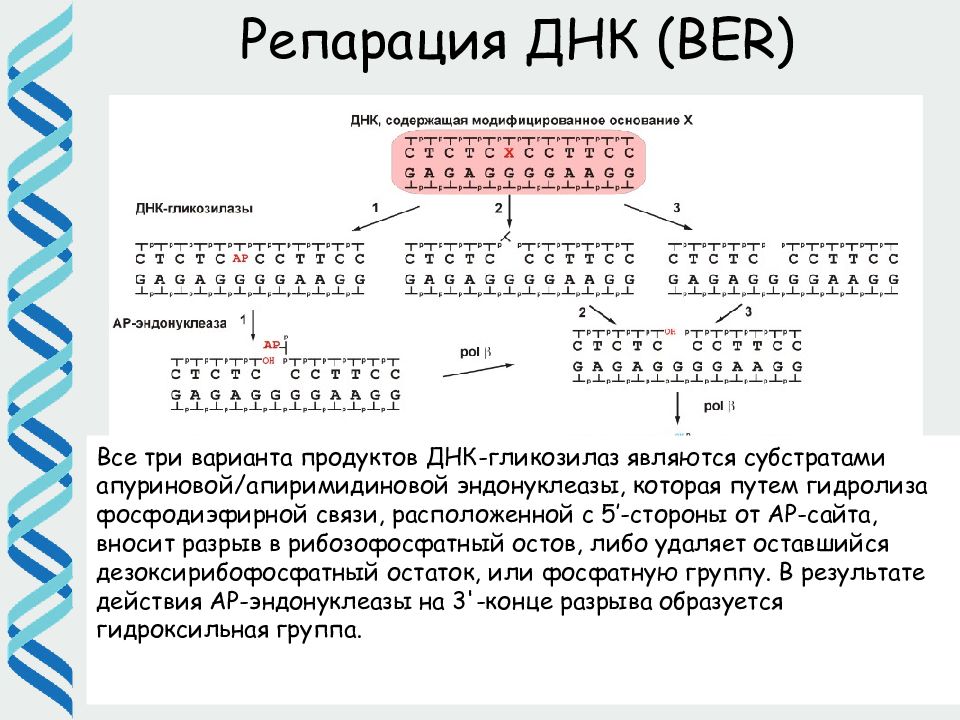
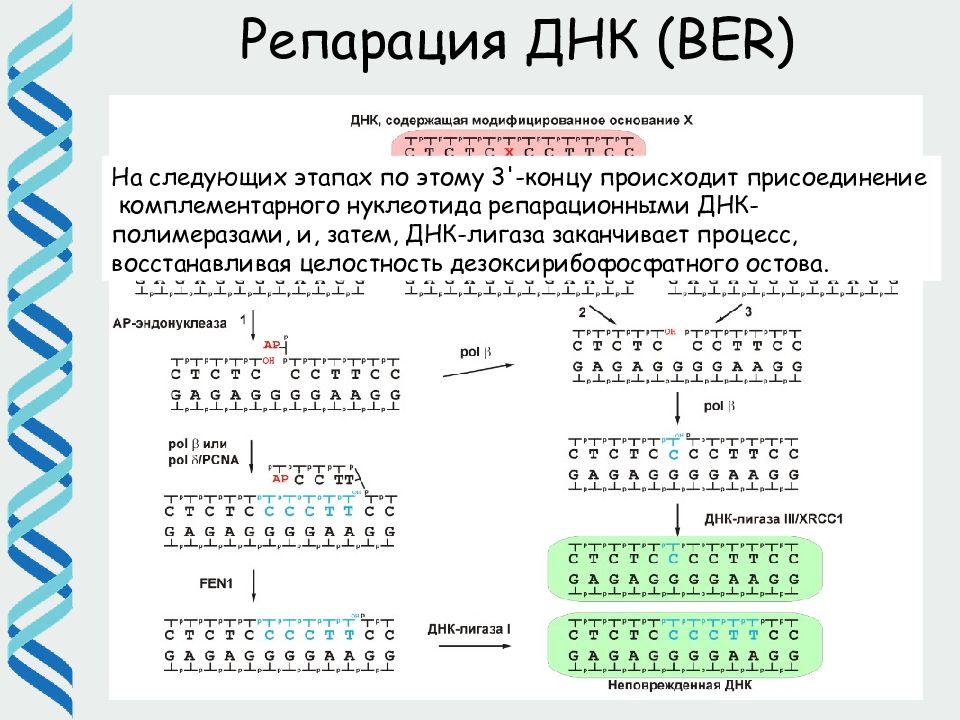
32 Репарация ДНК (BER) Все три варианта продуктов ДНК-гликозилаз являются субстратами апуриновой/апиримидиновой эндонуклеазы, которая путем гидролиза фосфодиэфирной связи, расположенной с 5′-стороны от АР-сайта, вносит разрыв в рибозофосфатный остов, либо удаляет оставшийся дезоксирибофосфатный остаток, или фосфатную группу. В результате действия АР-эндонуклеазы на 3'-конце разрыва образуется гидроксильная группа.
Слайд 33
33 Репарация ДНК (BER) На следующих этапах по этому 3'-концу происходит присоединение комплементарного нуклеотида репарационными ДНК-полимеразами, и, затем, ДНК-лигаза заканчивает процесс, восстанавливая целостность дезоксирибофосфатного остова.
Слайд 34
34 Репарация ДНК (NIR) Некоторые AP эндонуклеазы (например, APE1) могут работать как сенсор повреждений. Помимо AP сайтов они узнают ряд других повреждений, в том числе окисленные основания. Эта активность важна для альтернативного пути репарации единичных повреждений ( nucleotide incision repair, NIR). Преимущество: нет апуринового сайта, который сам по себе может быть опасным для клетки
Слайд 38
38 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
Слайд 39
39 Репарация ДНК ( NER) Используется для коррекции «серьезных» повреждений, которые блокируют репликацию (например, у человека – тиминовые димеры). 1. Специальные белки узнают поврежденные участки ДНК и привлекают специальные нуклеазы, которые вносят разрывы на некотором расстоянии перед повреждением и на некотором расстоянии после него. 2. Фрагмент ДНК, содержащий повреждение, удаляется, и образовавшаяся брешь застраивается ДНК полимеразой
Слайд 41
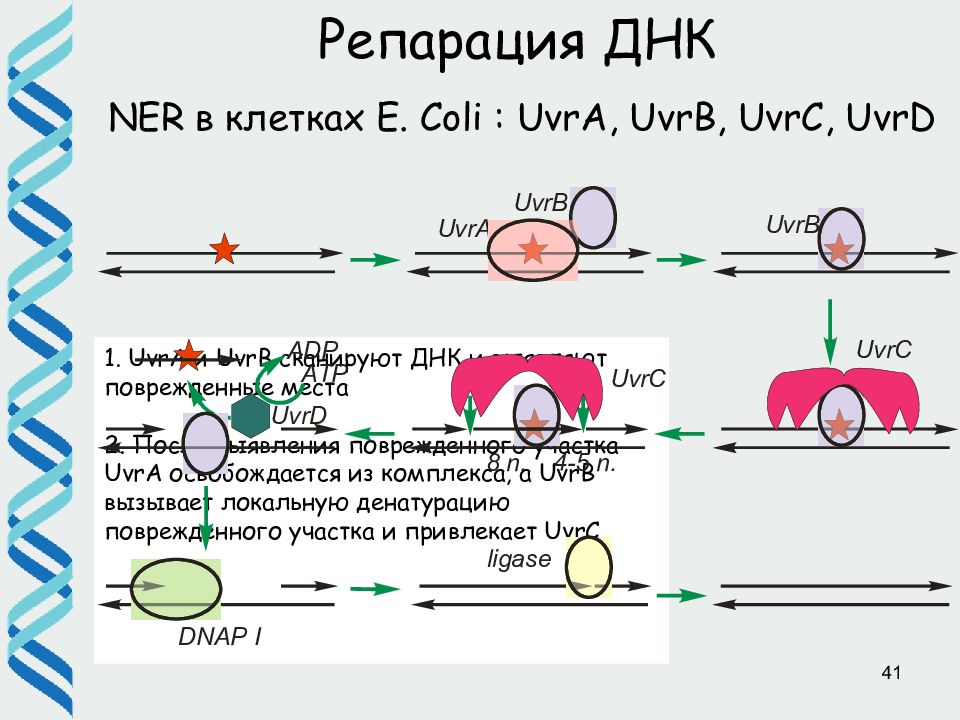
41 Репарация ДНК NER в клетках E. Coli : UvrA, UvrB, UvrC, UvrD 1. UvrA и UvrB сканируют ДНК и выявляют поврежденные места 2. После выявления поврежденного участка UvrA освобождается из комплекса, а UvrB вызывает локальную денатурацию поврежденного участка и привлекает UvrC
Слайд 42
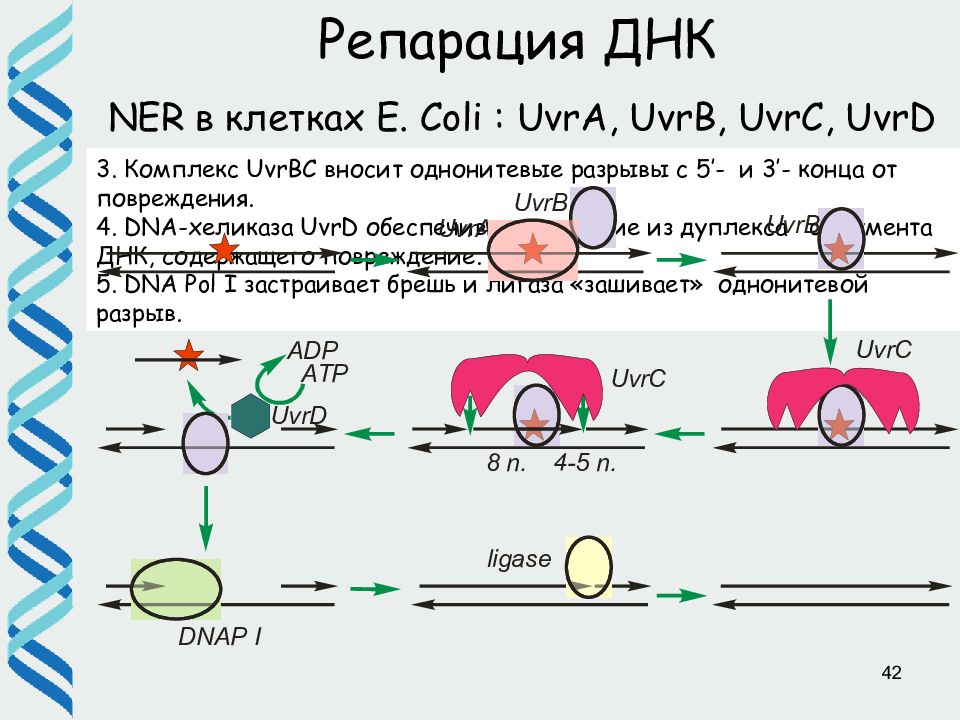
42 Репарация ДНК NER в клетках E. Coli : UvrA, UvrB, UvrC, UvrD 3. Комплекс UvrB С вносит однонитевые разрывы с 5 ’- и 3’- конца от повреждения. 4. DNA- хеликаза UvrD обеспечивает удаление из дуплекса фрагмента ДНК, содержащего повреждение. 5. DNA Pol I застраивает брешь и лигаза «зашивает» однонитевой разрыв.
Слайд 43
43 Репарация ДНК NER в клетках E. Coli : UvrA, UvrB, UvrC, UvrD Практически вся эксцизионная репарация в клетках E.coli проходит за счет UvrABC В 99% случаев длина «заплатки» 12 н.о. – ремонт короткими «заплатками» 1% – примерно 1500 н.о. (бывает > 9000 н.о.) – ремонт длинными «заплатками»
Слайд 44
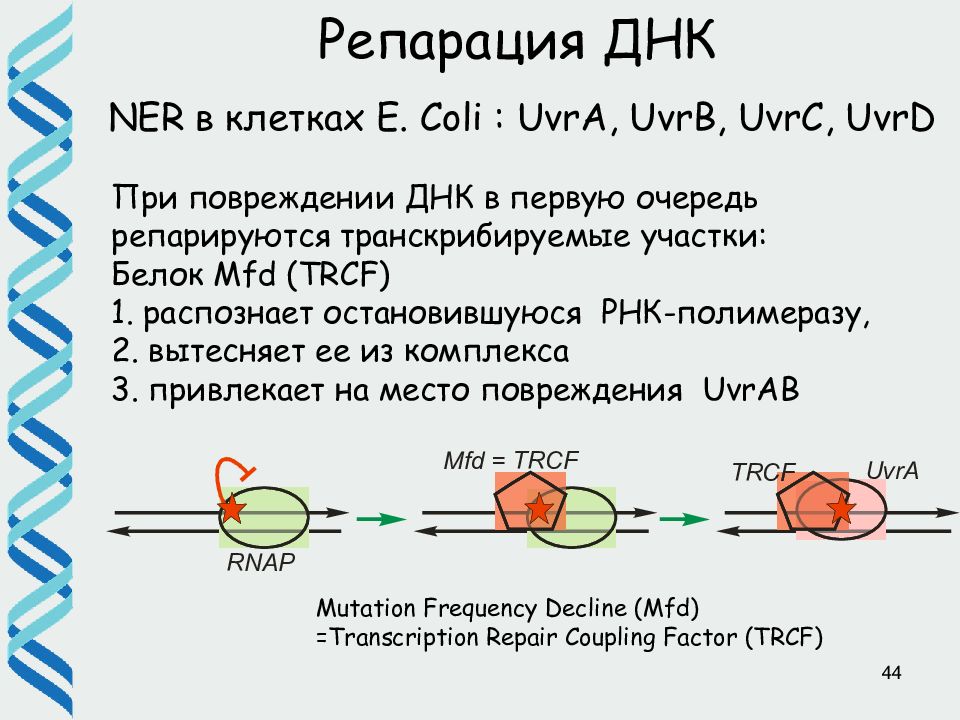
44 Репарация ДНК NER в клетках E. Coli : UvrA, UvrB, UvrC, UvrD Mutation Frequency Decline (Mfd) =Transcription Repair Coupling Factor (TRCF) При повреждении ДНК в первую очередь репарируются транскрибируемые участки: Белок Mfd ( TRCF) 1. распознает остановившуюся РНК-полимеразу, 2. вытесняет ее из комплекса 3. привлекает на место повреждения UvrAB
Слайд 45
45 Репарация ДНК NER в эукариотических клетках Основной принцип NER в клетках эукариот такой же, как и в бактериальных: репарируются крупные повреждения, полученные под действием УФ-света, сшивающих агентов, химических канцерогенов, путем вырезания фрагмента ДНК Два пути NER : 1. Система глобальной геномной репарации (GG-NER) 2. Репарация ДНК, связанная с транскрипцией ( TC-NER) Нарушения работе NER ведут к серьезным заболеваниям. Пигментная ксеродерма ( Xeroderma Pigmentosum ) (XP) рецессивное заболевание, семь генов (XPA-XPG) 1-4 случая на 1 000 000 человек чувствительность к солнечному свету, особенно к УФ, дефекты в работе ранних этапов NER
Слайд 46
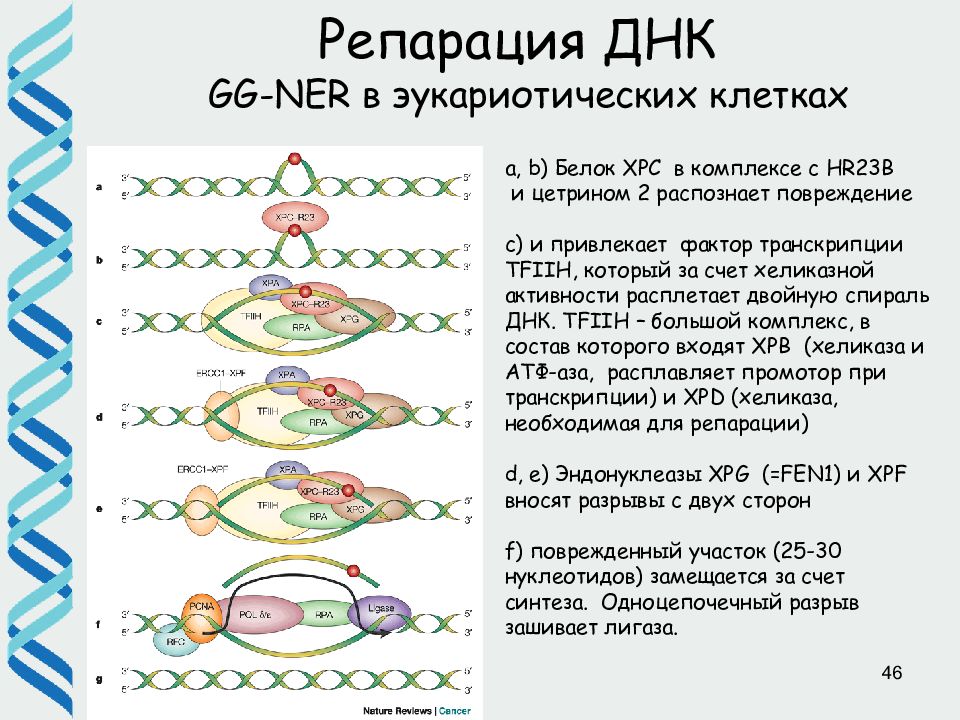
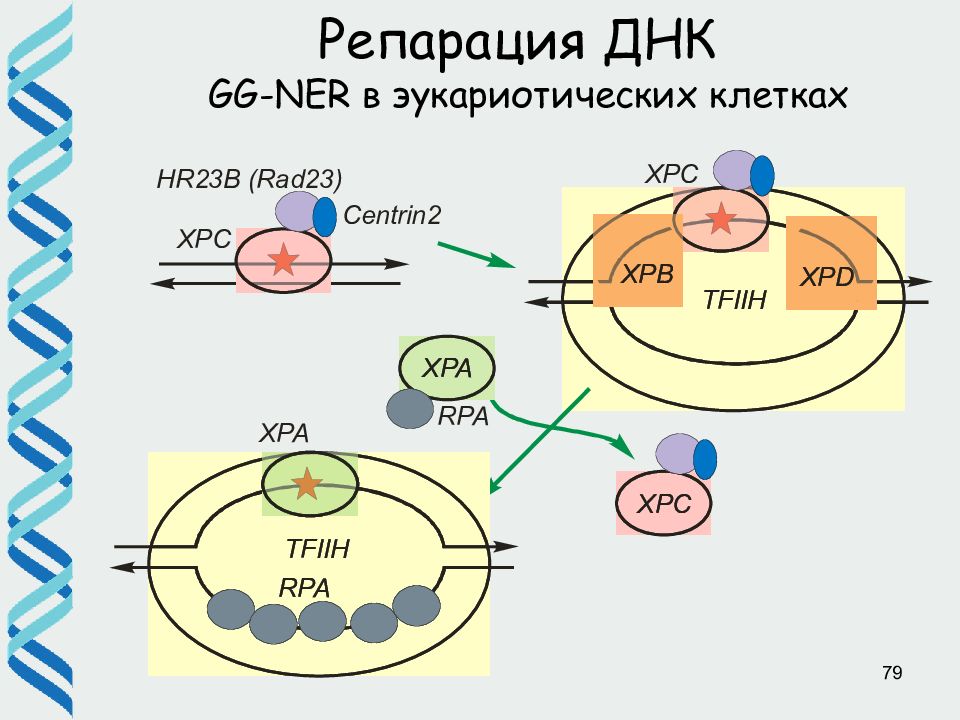
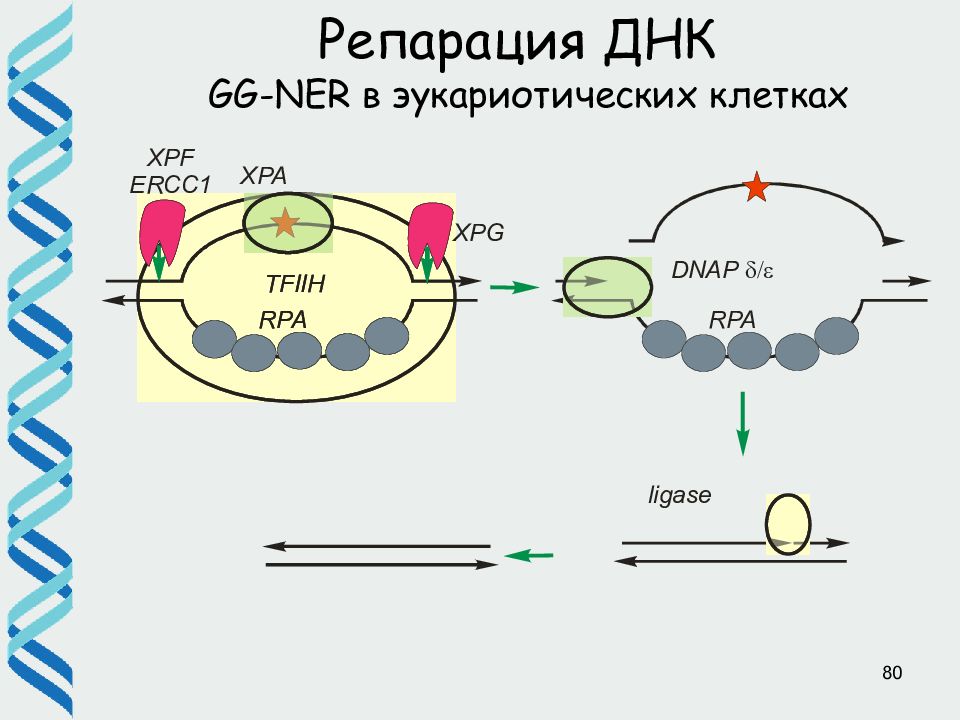
46 Репарация ДНК GG-NER в эукариотических клетках a, b) Белок XPC в комплексе c HR23B и цетрином 2 распознает повреждение c) и привлекает фактор транскрипции TFIIH, который за счет хеликазной активности расплетает двойную спираль ДНК. TFIIH – большой комплекс, в состав которого входят XPB (хеликаза и АТФ-аза, расплавляет промотор при транскрипции) и XPD (хеликаза, необходимая для репарации) d, e) Эндонуклеазы XPG (= FEN1) и XPF вносят разрывы с двух сторон f) поврежденный участок (25-30 нуклеотидов) замещается за счет синтеза. Одноцепочечный разрыв зашивает лигаза.
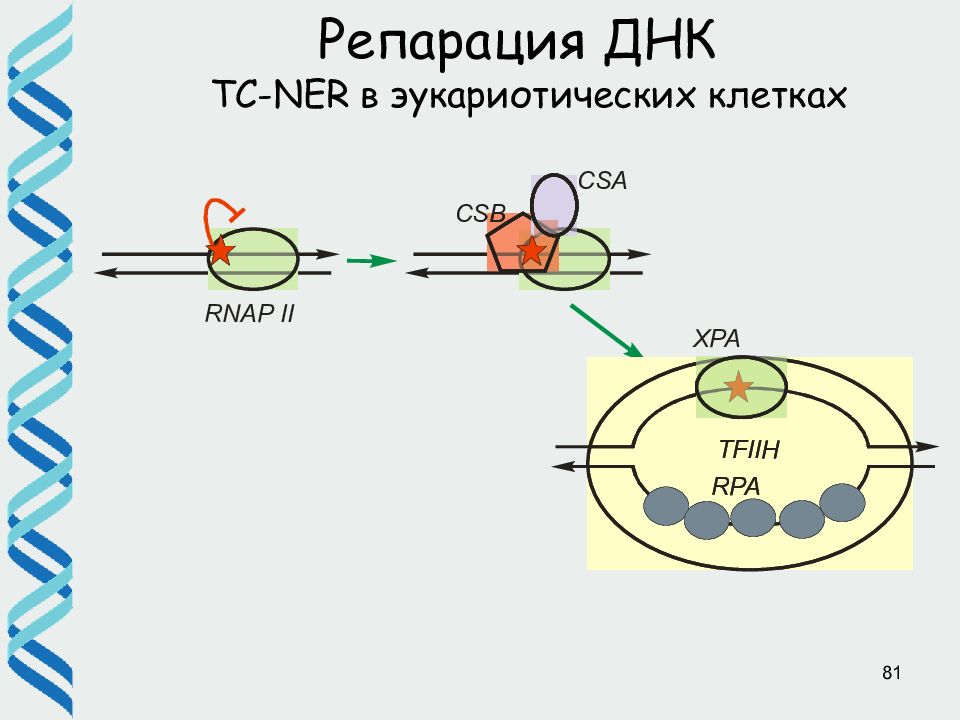
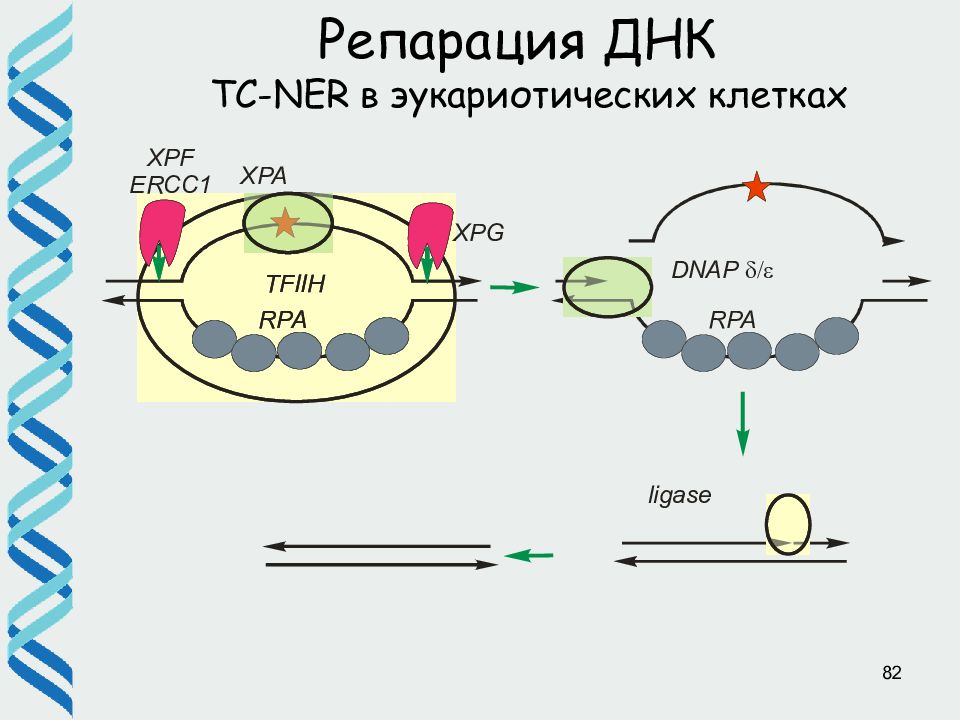
Слайд 47
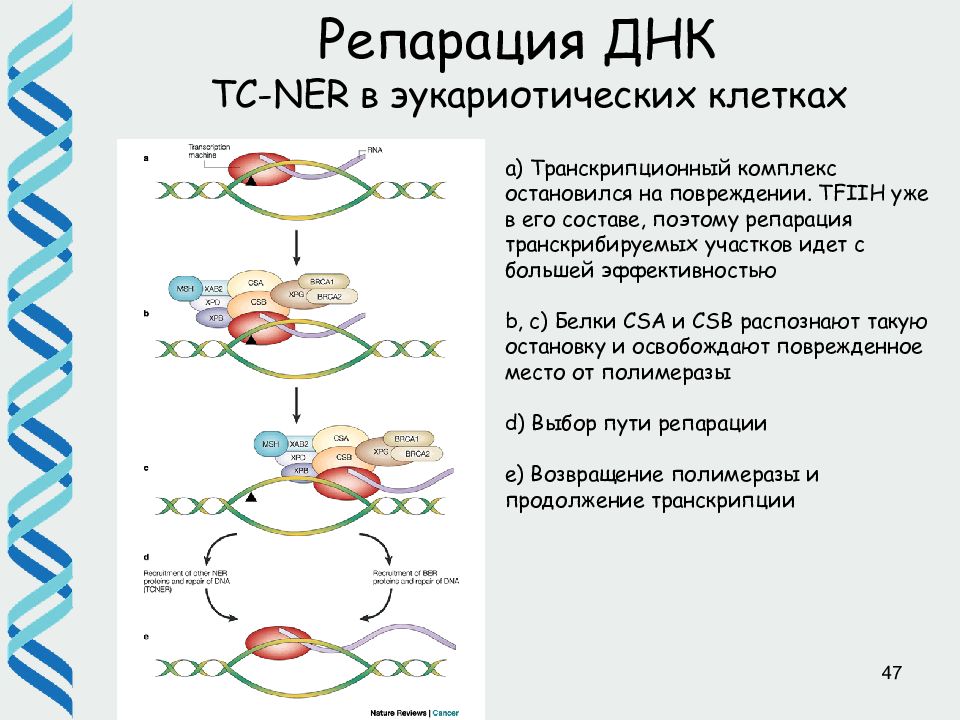
47 Репарация ДНК TC-NER в эукариотических клетках a) Транскрипционный комплекс остановился на повреждении. TFIIH уже в его составе, поэтому репарация транскрибируемых участков идет с большей эффективностью b, c) Белки CSA и CSB распознают такую остановку и освобождают поврежденное место от полимеразы d) Выбор пути репарации e) Возвращение полимеразы и продолжение транскрипции
Слайд 48
48 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
Слайд 49
49 Репарация ДНК ( MMR) ДНК полимеразы (даже те, у которых есть корректирующая активность) все равно делают ошибки, которые надо исправлять Система репарации ошибок репликации должна 1. Быстро находить ошибки 2. Различать родительскую и новосинтезированную цепь с тем, чтобы в неспаренном участке заменить ошибочно включенный нуклеотид
Слайд 50
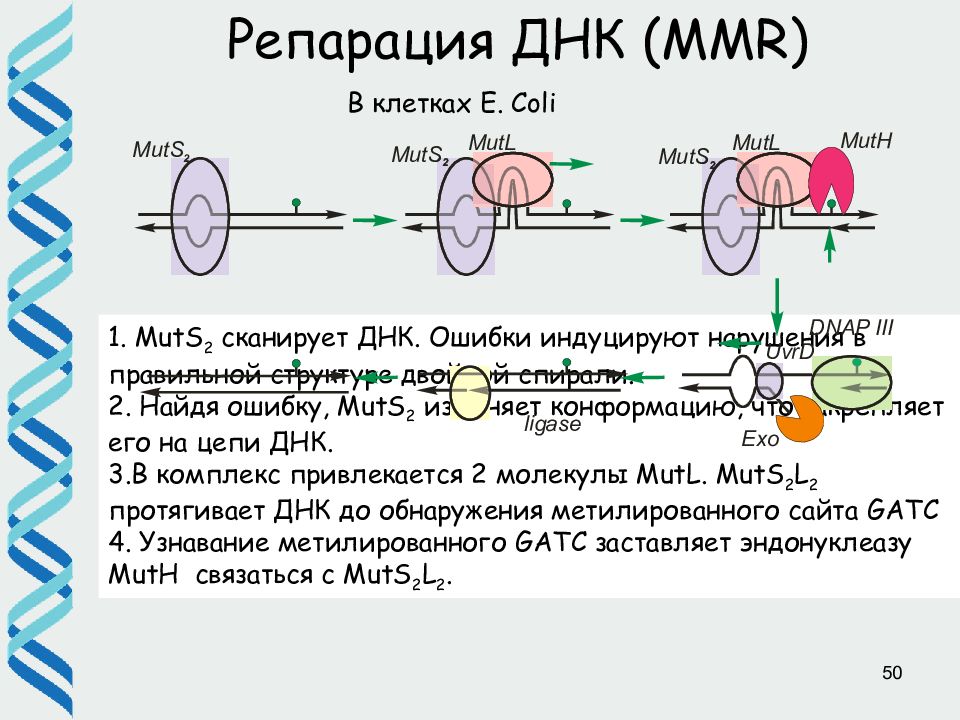
50 Репарация ДНК ( MMR) 1. MutS 2 сканирует ДНК. Ошибки индуцируют нарушения в правильной структуре двойной спирали. 2. Найдя ошибку, MutS 2 изменяет конформацию, что закрепляет его на цепи ДНК. 3.В комплекс привлекается 2 молекулы MutL. MutS 2 L 2 протягивает ДНК до обнаружения метилированного сайта GATC 4. Узнавание метилированного GATC заставляет эндонуклеазу MutH связаться с MutS 2 L 2. В клетках E. Coli
Слайд 51
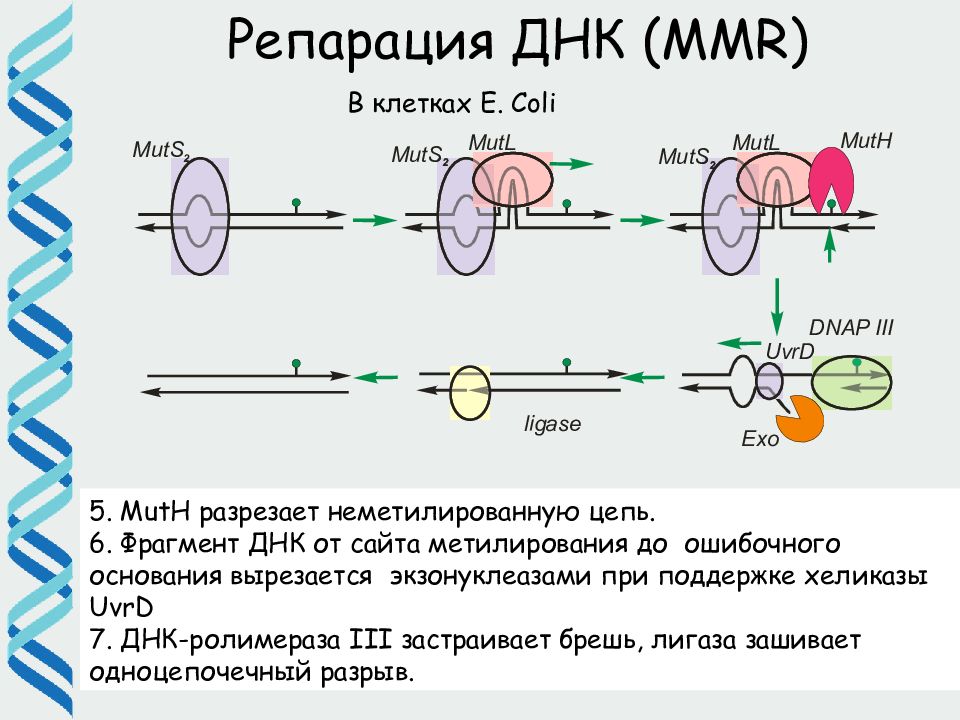
51 Репарация ДНК ( MMR) 5. MutH разрезает неметилированную цепь. 6. Фрагмент ДНК от сайта метилирования до ошибочного основания вырезается экзонуклеазами при поддержке хеликазы UvrD 7. ДНК-ролимераза III застраивает брешь, лигаза зашивает одноцепочечный разрыв. В клетках E. Coli
Слайд 52
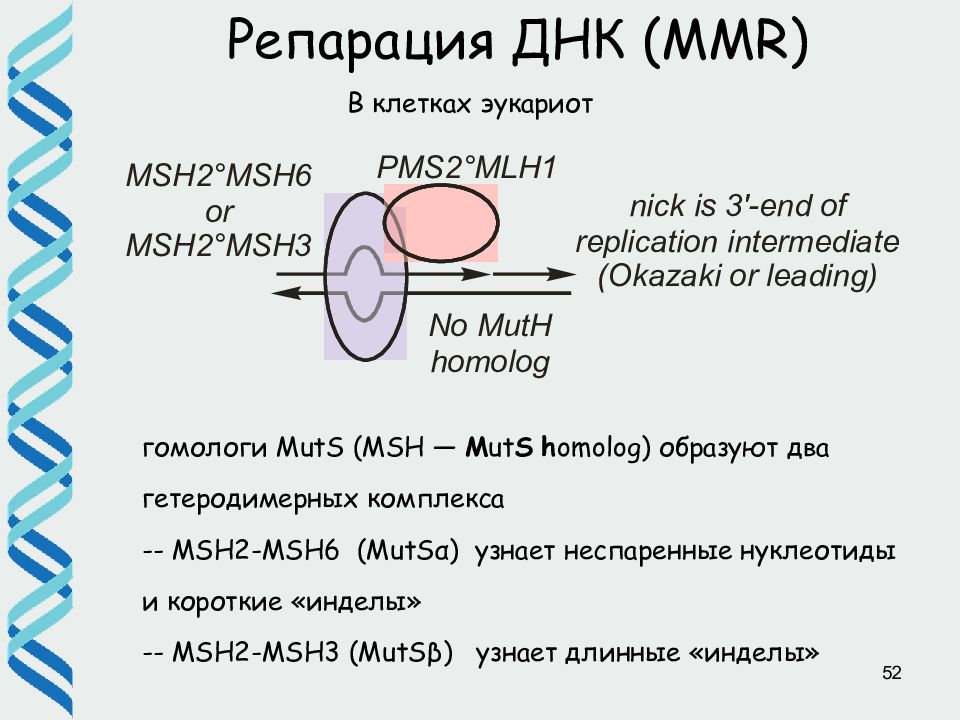
52 Репарация ДНК ( MMR) В клетках эукариот гомологи MutS (MSH — M ut S h omolog) образуют два гетеродимерных комплекса -- MSH2-MSH6 (MutSα) узнает неспаренные нуклеотиды и короткие «инделы» -- MSH2-MSH3 (MutSβ) узнает длинные «инделы»
Слайд 53
53 Репарация ДНК ( MMR) Эксперименты по связыванию с ДНК in vitro и репарации гетеродуплексов in vivo показали, что MMR узнает все комбинации неспаренных оснований, кроме C:C, а также короткие <4 п.н. делеции и инсерции («инделы») Неправильные пары G:T and A:C и инсерции/делеции в 1 п. особенно хорошо узнаются. Эти нарушения являются наиболее частыми ошибками ДНК-полимераз
Слайд 54
54 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
Слайд 55
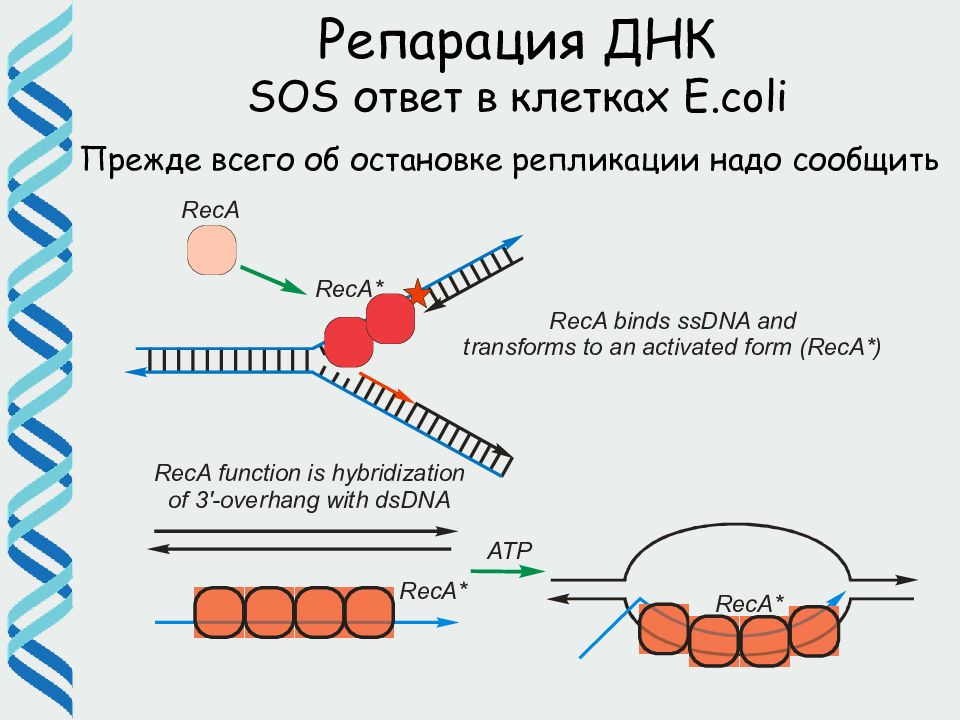
Прежде всего об остановке репликации надо сообщить Rec A Связывается с однонитевой ДНК и образует ДНК-белковые филаменты Однонитевые участки ДНК образуются при остановке репликативных вилок Участвует в рекомбинации и индукции SOS ответа Lex A (репрессор) Мастер-регулятор транскрипции генов, кодирующих участвующие в репарации повреждений ДНК белки (31 ген или более) Димеры Lex A связываются с SOS боксами (20 п.н. консенсусы) в операторах генов репарации и ингибируют транскрипцию Репарация ДНК SOS ответ в клетках E.coli
Слайд 56
Прежде всего об остановке репликации надо сообщить Репарация ДНК SOS ответ в клетках E.coli
Слайд 57
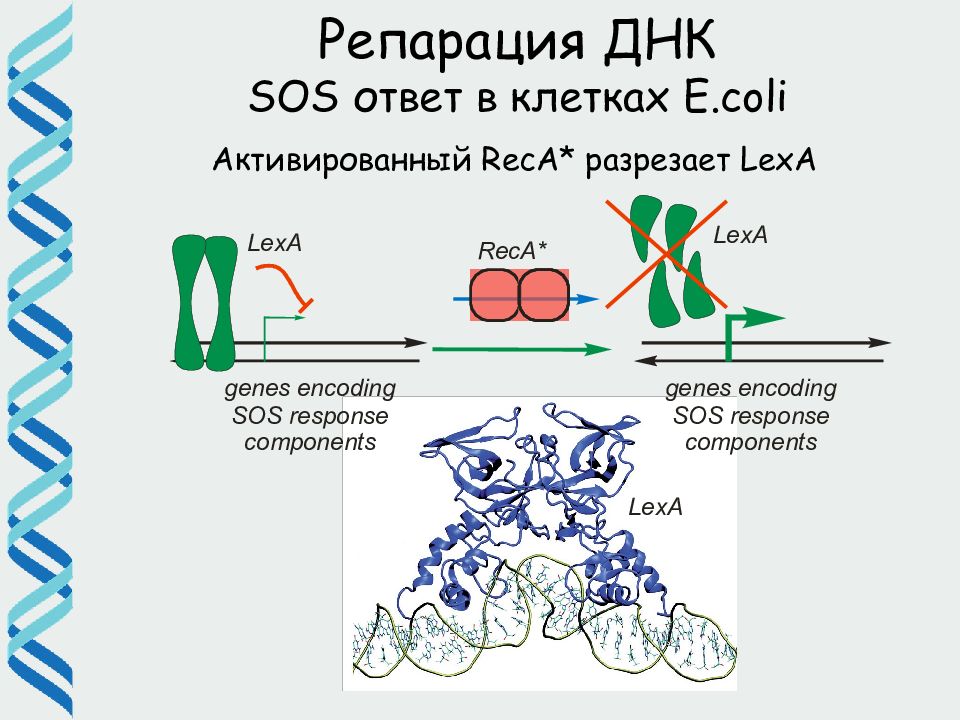
Репарация ДНК SOS ответ в клетках E.coli Активированный RecA* разрезает LexA
Слайд 58
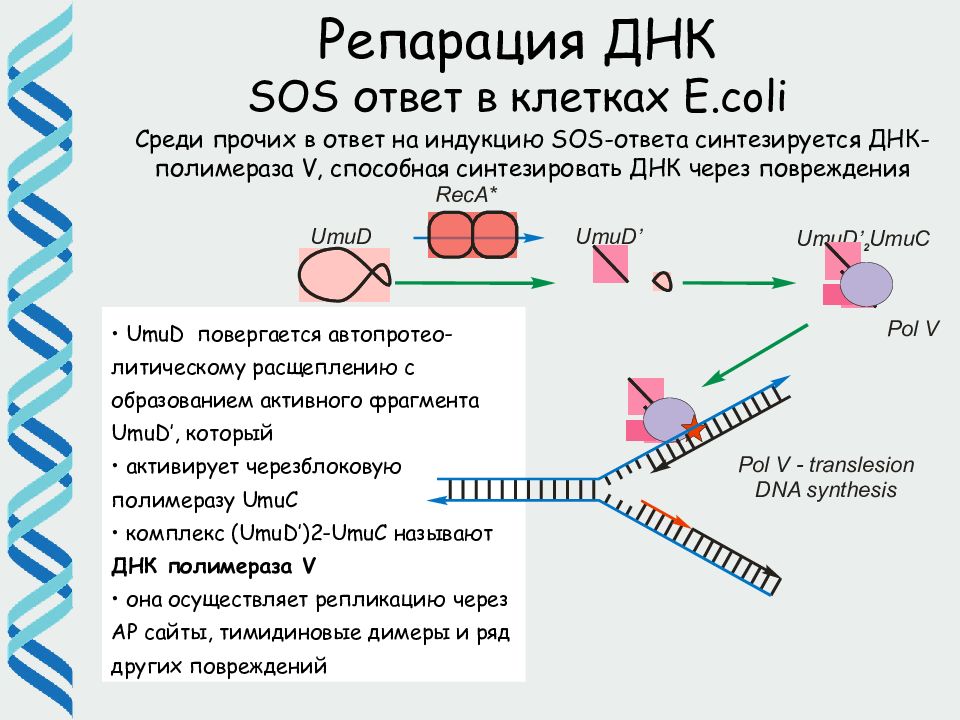
Репарация ДНК SOS ответ в клетках E.coli Среди прочих в ответ на индукцию SOS -ответа синтезируется ДНК-полимераза V, способная синтезировать ДНК через повреждения • UmuD повергается автопротео-литическому расщеплению с образованием активного фрагмента UmuD ’, который • активирует черезблоковую полимеразу UmuC • комплекс (UmuD ’ )2-UmuC называют ДНК полимераза V • она осуществляет репликацию через AP сайты, тимидиновые димеры и ряд других повреждений
Слайд 59
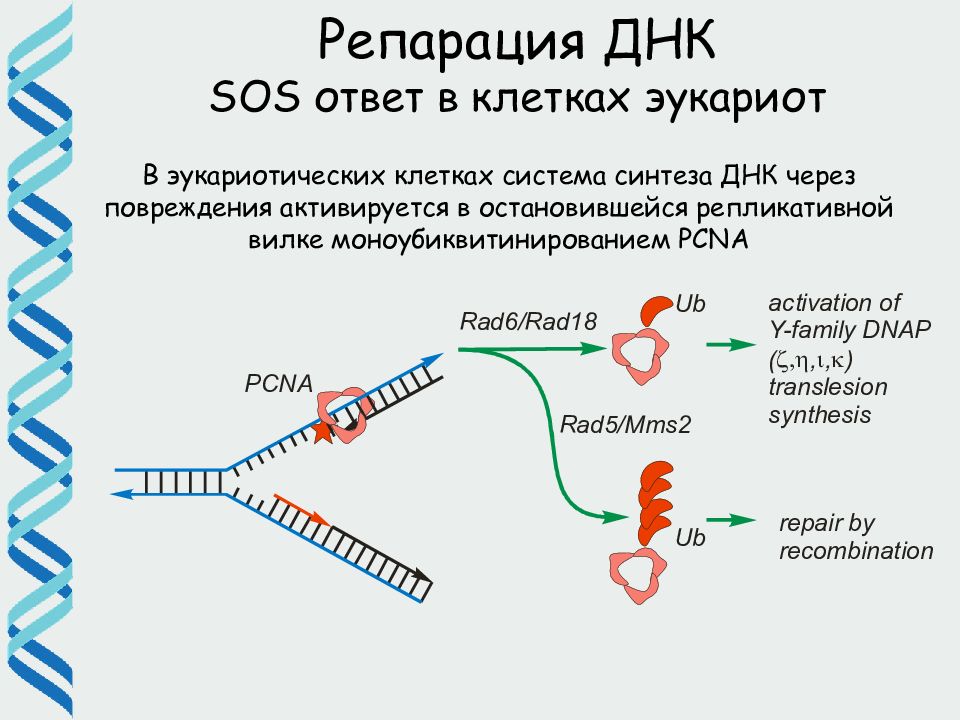
Репарация ДНК SOS ответ в клетках эукариот В эукариотических клетках система синтеза ДНК через повреждения активируется в остановившейся репликативной вилке моноубиквитинированием PCNA
Слайд 61
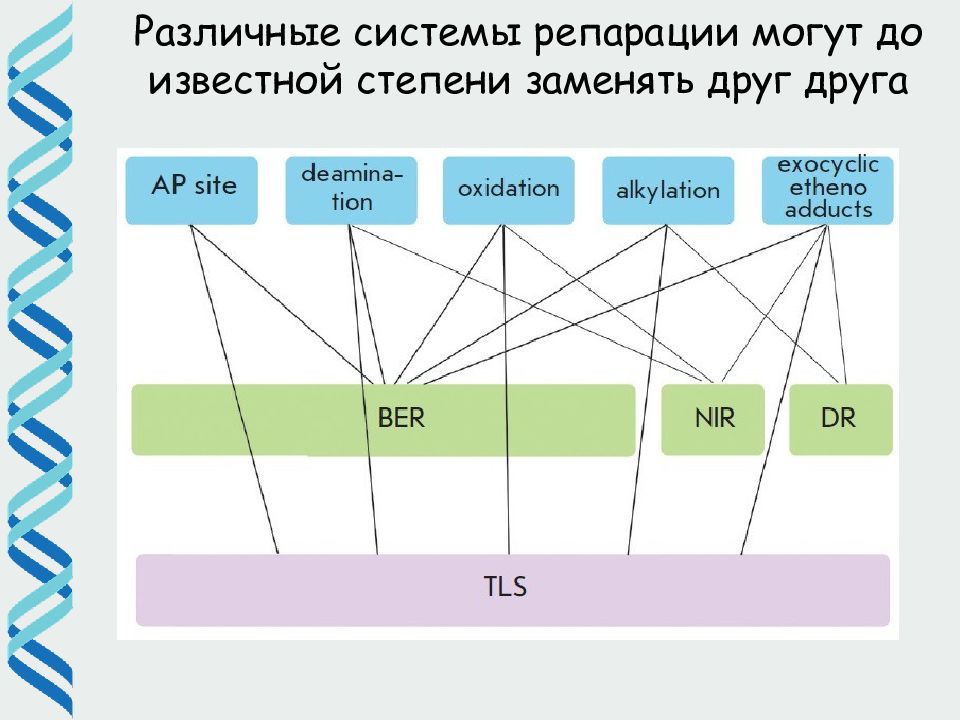
Различные системы репарации могут до известной степени заменять друг друга
Слайд 62
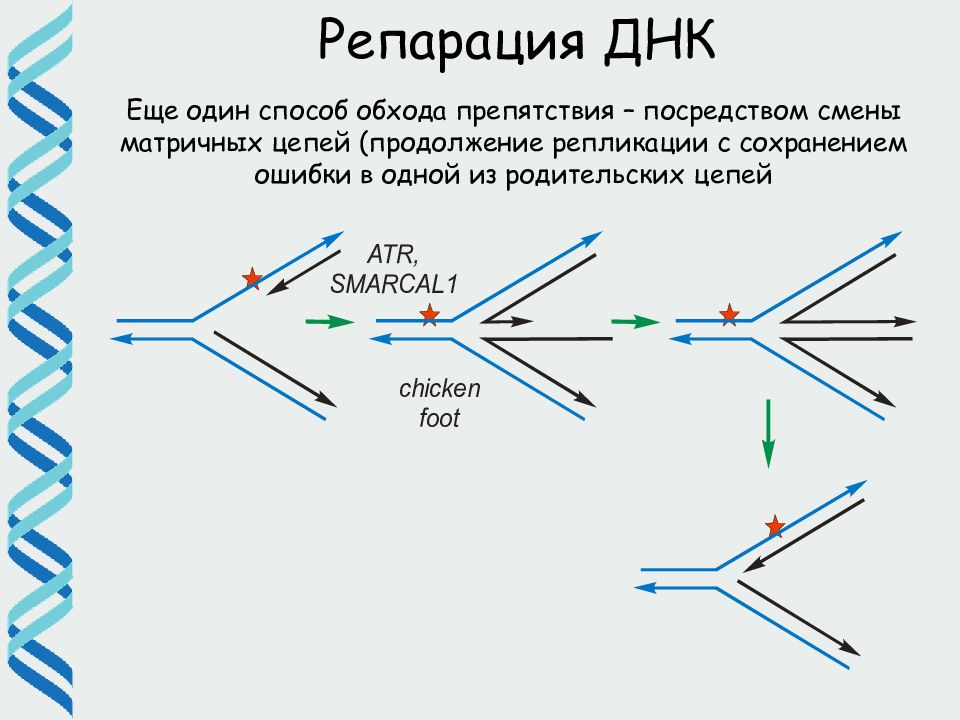
Репарация ДНК Еще один способ обхода препятствия – посредством смены матричных цепей ( продолжение репликации с сохранением ошибки в одной из родительских цепей
Слайд 63
63 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
Слайд 64
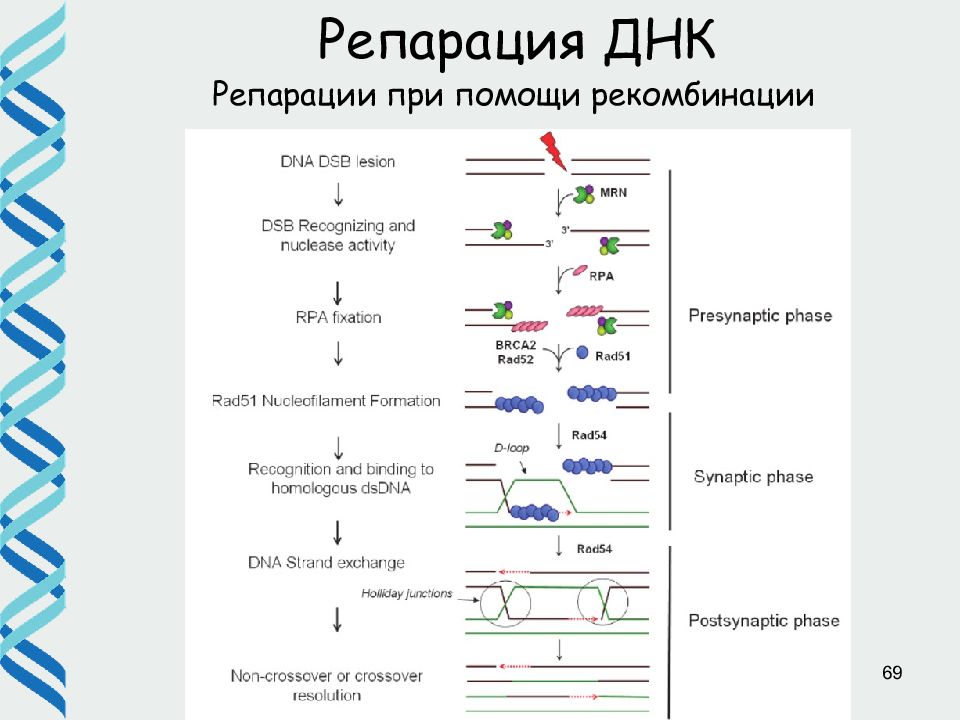
64 Репарации при помощи рекомбинации Репарация ДНК Для репарации двунитевых разрывов с использованием системы гомологичной рекомбинации необходимы Донор гомологии (например гомологичная хромосома или сестринская хроматида) Белок, облегчающий инвазию цепи, и другие компоненты системы гомологичной рекомбинации
Слайд 65
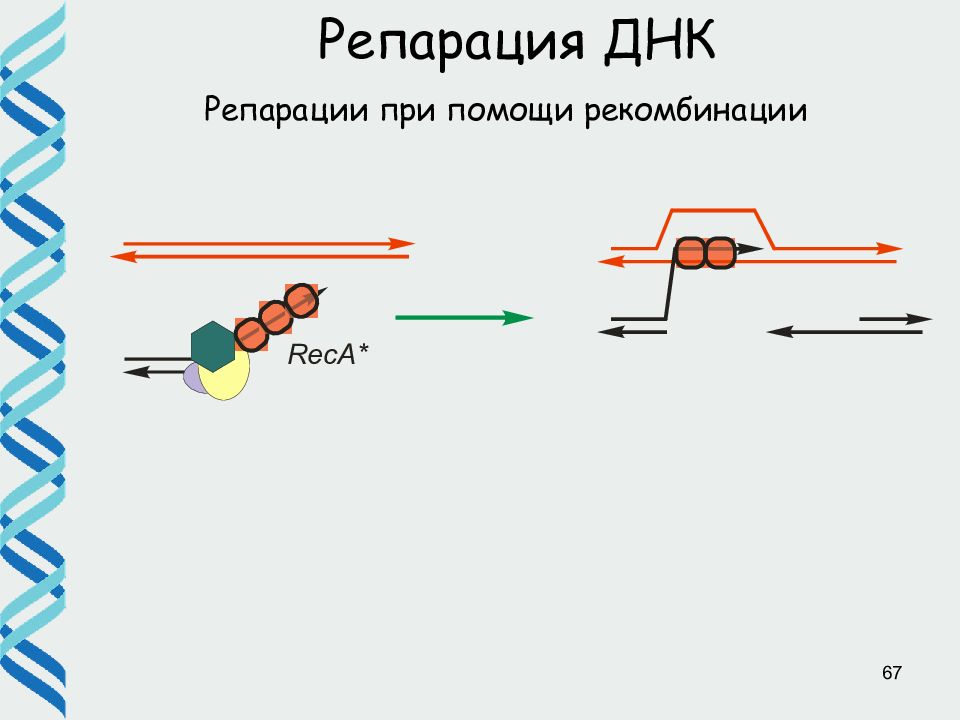
65 Репарация ДНК Репарации при помощи рекомбинации Процессирование концов экзонуклеазами; Создание выступающих 3 ’ - концов Инвазия 3 ’ - конца первой цепи Инвазия 3 ’ - конца второй цепи и репаративный синтез Миграция ветвей с последующим образованием классической структуры Холидея
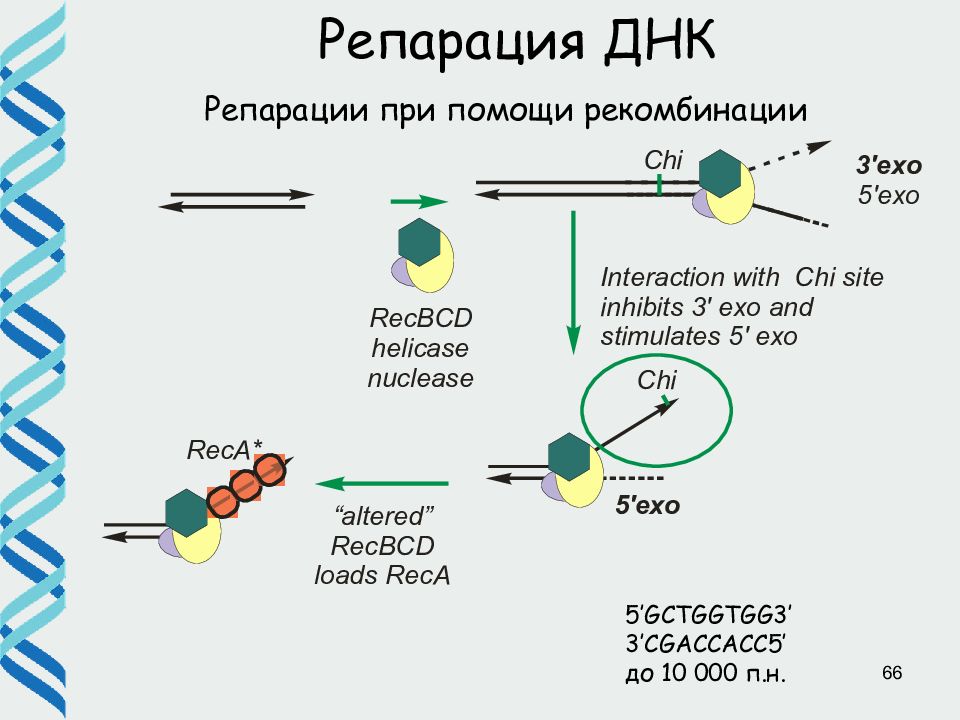
Слайд 66
66 Репарации при помощи рекомбинации Репарация ДНК 5’GCTGGTGG3’ 3’CGACCACC5’ до 10 000 п.н.
Слайд 68
68 Репарации при помощи рекомбинации Репарация ДНК 22.13 DS SS участок гомологии
Слайд 70
70 Репарация ДНК Прямое исправление повреждений (Direct reversal) (2 и >) 2. Эксцизионная репарация оснований (Base excision repair (BER)) (15) 3. Эксцизионная репарация нуклеотидов (Nucleotides excision repair (NER)) (28) 4. Репарация неправильно спаренных нуклеотидов (Mismatch repair (MMR)) (11) 5. Синтез через повреждения (СОС-ответ) (Trans-lesion synthesis (SOS-response)) 6. Репарация при помощи рекомбинации (Repair via recombination) (14) 7. Репарация двойных разрывов (Double strand break repair) (5) Пути репарации ДНК
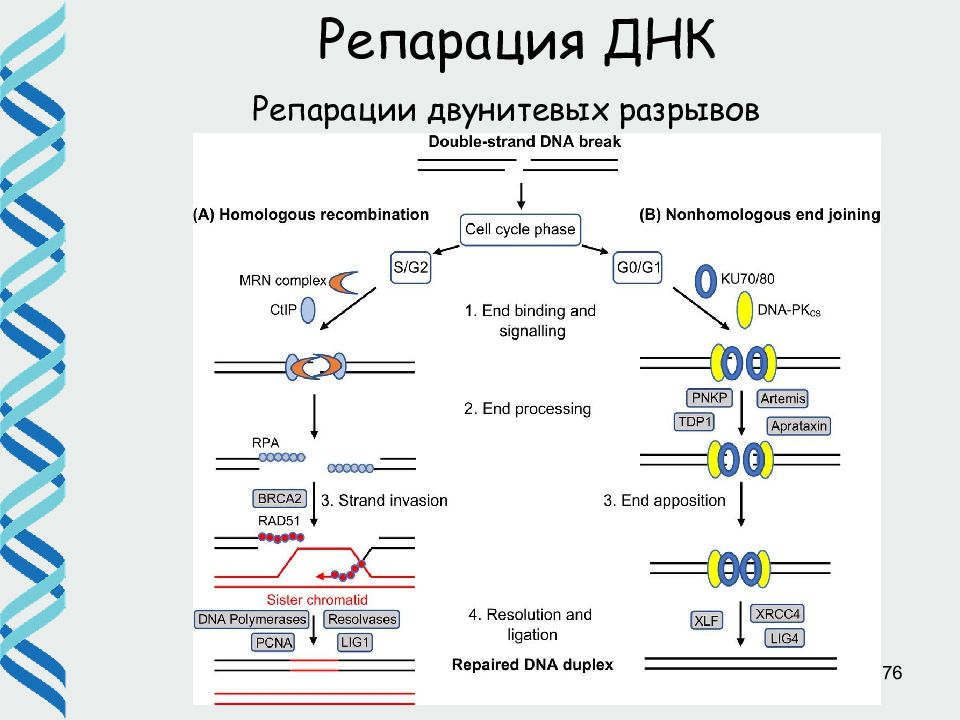
Слайд 71
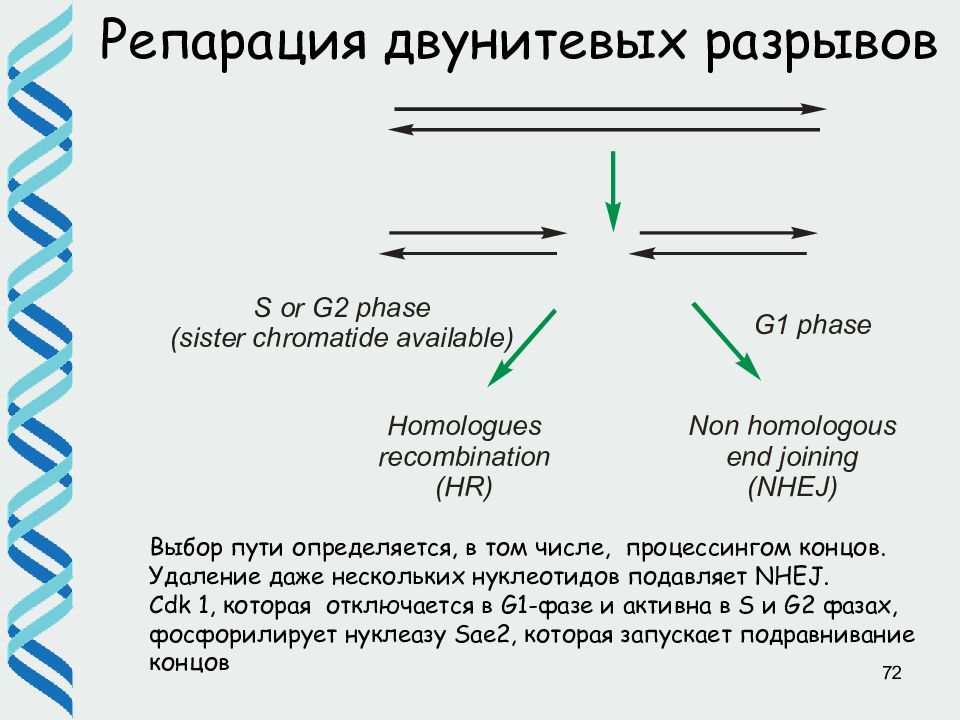
71 Репарация двунитевых разрывов Существует два основных пути репарации двунитевых разрывов: • гомологичная рекомбинация • негомологичное соединение концов ДНК Двунитевые разрывы в ДНК возникают : • под действием ионизирующего излучения • под действием некоторых химических агентов, в частности, ингибиторов ДНК топоизомеразы II
Слайд 72
72 Репарация двунитевых разрывов Выбор пути определяется, в том числе, процессингом концов. Удаление даже нескольких нуклеотидов подавляет NHEJ. Cdk 1, которая отключается в G1-фазе и активна в S и G2 фазах, фосфорилирует нуклеазу Sae2, которая запускает подравнивание концов
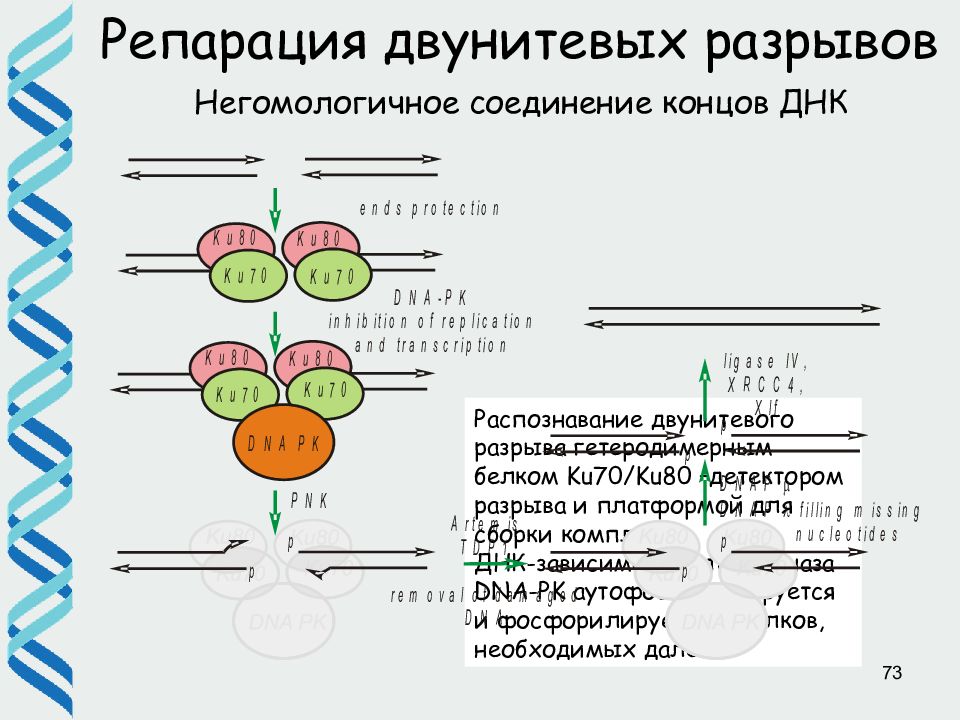
Слайд 73
73 Репарация двунитевых разрывов Негомологичное соединение концов ДНК Распознавание двунитевого разрыва гетеродимерным белком Ku70/Ku80 –детектором разрыва и платформой для сборки комплекса ДНК-зависимая протеинкиназа DNA-PK аутофосфорилируется и фосфорилирует ряд белков, необходимых далее
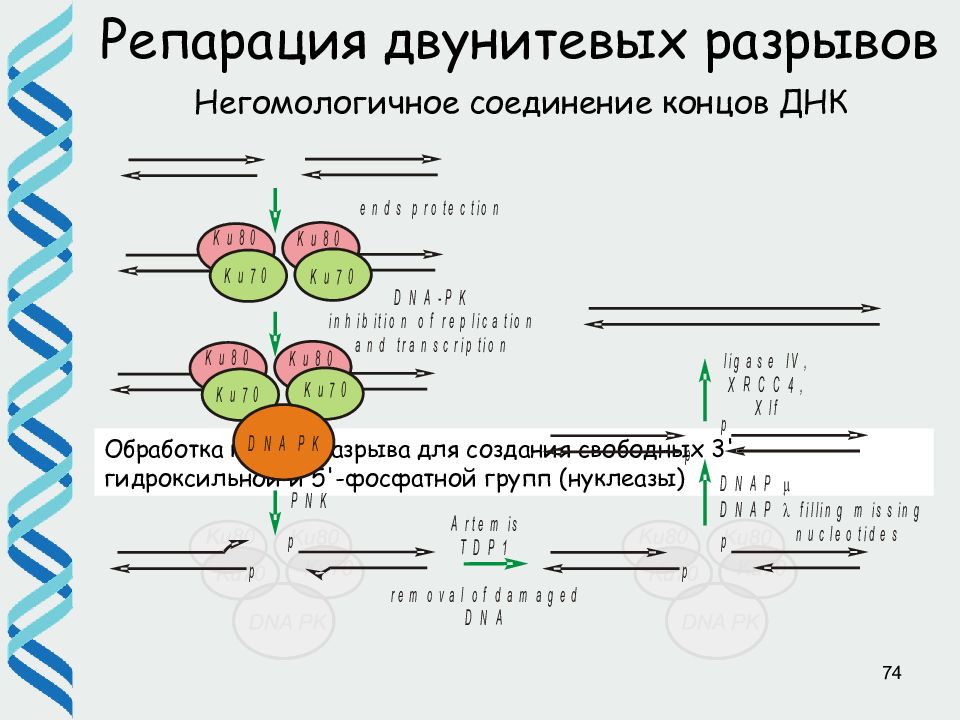
Слайд 74
74 Репарация двунитевых разрывов Негомологичное соединение концов ДНК Обработка концов разрыва для создания свободных 3'-гидроксильной и 5'-фосфатной групп (нуклеазы)
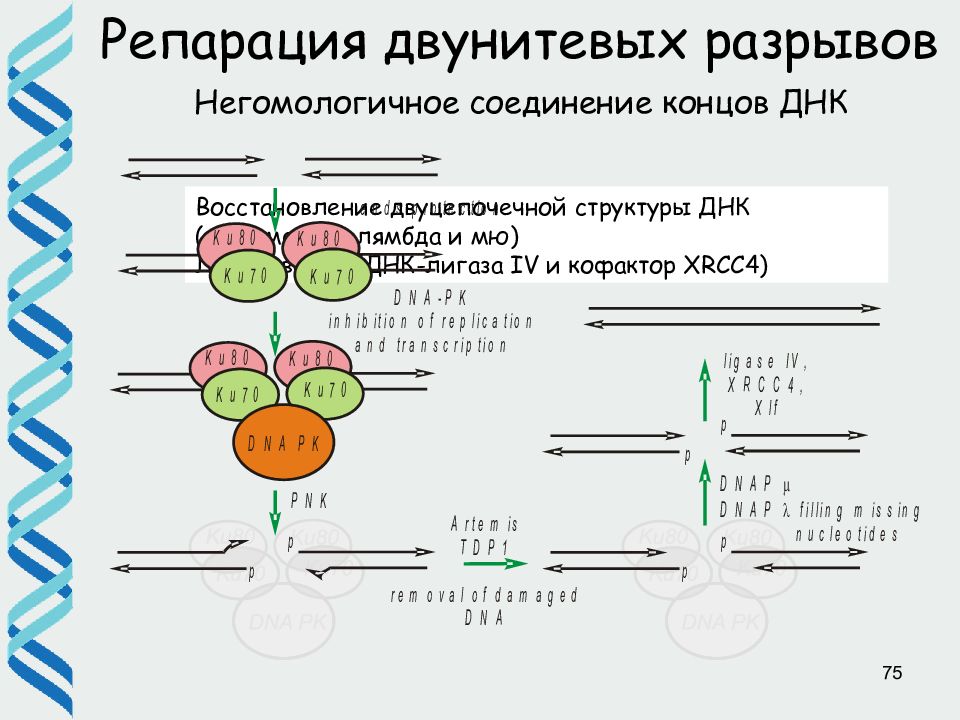
Слайд 75
75 Репарация двунитевых разрывов Негомологичное соединение концов ДНК Восстановление двуцепочечной структуры ДНК (полимеразы лямбда и мю) Лигирование (ДНК-лигаза IV и кофактор XRCC4)