Слайд 2: Биоинформатика
Современная междисциплинарная наука, которая использует современные компьютерные технологии для решения биологических проблем Сбор, хранение, анализ и интерпритация биологической информации Молекулярное моделирование Открытие генов Определение функции генов Определение вторичной, третичной структуры белков и РНК Сравнение белков, ДНК, РНК …..
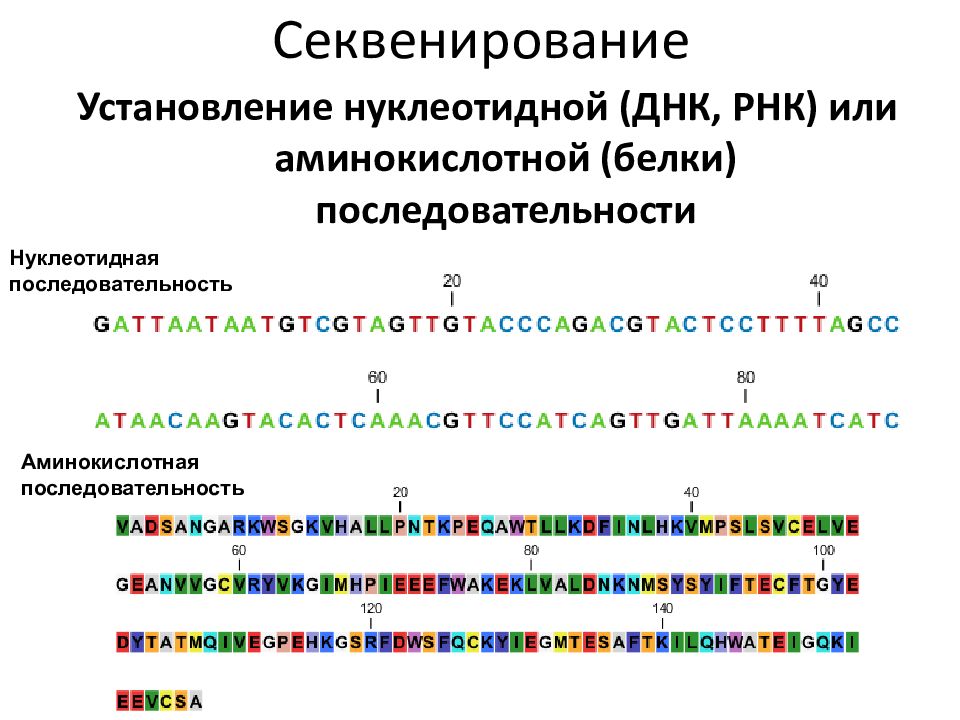
Слайд 3: Секвенирование
Установление нуклеотидной (ДНК, РНК) или аминокислотной (белки) последовательности Аминокислотная последовательность Нуклеотидная последовательность
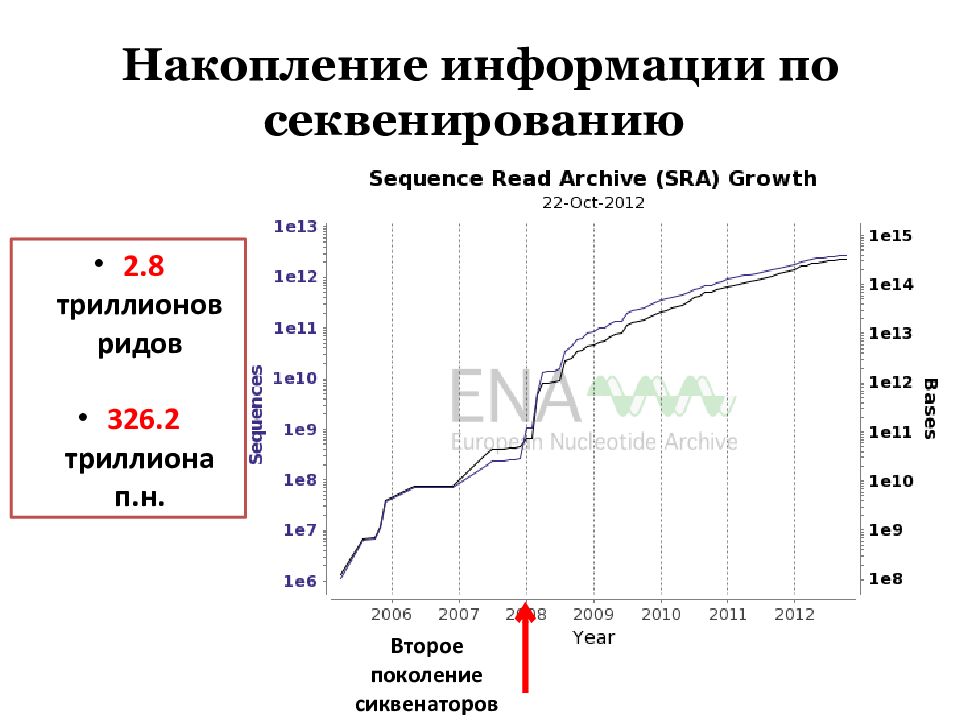
Слайд 4: Накопление информации по секвенированию
2.8 триллионов ридов 326.2 триллиона п.н. Второе поколение сиквенаторов
Слайд 5
Биоинформатика включает в себя: базы данных, в которых хранится биологическая информация (банки данных) набор инструментов для анализа тех данных, которые лежат в таких базах правильное применение компьютерных методов для правильного решения биологических задач
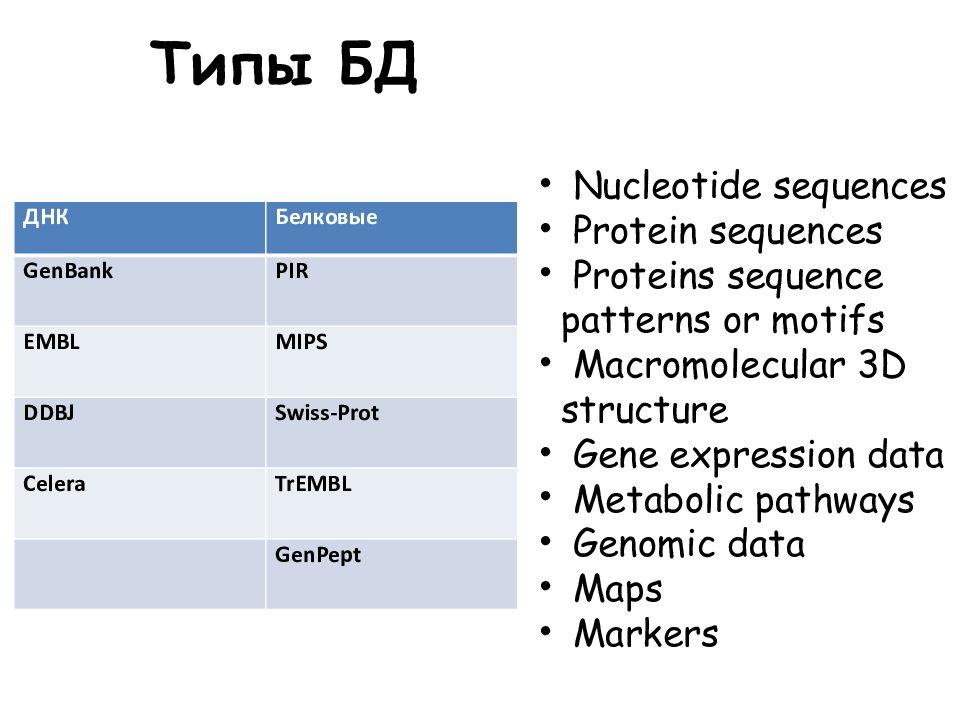
Слайд 7: Типы БД
Nucleotide sequences Protein sequences Proteins sequence patterns or motifs Macromolecular 3D structure Gene expression data Metabolic pathways Genomic data Maps Markers ДНК Белковые GenBank PIR EMBL MIPS DDBJ Swiss-Prot Celera TrEMBL GenPept
Слайд 8: Типы БД
Архивные Курируемые Производные Интегрированные Локальные Частные Общедоступные Типы БД
Слайд 9: Архивные БД
Архивные базы данных, это большая свалка, куда любой может поместить все, что захочет. К таким базам относятся: GeneBank & EMBL – здесь хранятся первичные последовательности PDB – пространственные структуры белков, и многое другое.
Слайд 10: Курируемые БД
За достоверность отвечает владельцы базы данных. Информацию из архивных баз данных отбирают эксперты, проверяя достоверность информации – что записано в этих последовательностях, какие есть экспериментальные основания для того, чтобы считать, что эти последовательности выполняют ту или иную функцию. К базам данных такого типа относятся: Swiss- Prot – наиболее качественная база данных, содержащая аминокислотные последовательности белков KEGG – информация о метаболизме FlyBase – информация о Drosophila COG – информация об ортологичных генах прокариот KOG - информация об ортологичных генах эукариот TAIL – информация о Arabidopsis thaliana
Слайд 11: Производные БД
Такие базы получаются в результате обработки данных из архивных и курируемых баз данных. Сюда входит: SCOP – База данных структурной классификации белков (описывается структура белков) PFAM – База данных по семействам белков GO (Gene Ontology) – Классификация генов (попытка создания набора терминов, упорядочивания терминологии, чтобы один ген не назывался по разному, и чтобы разным генам не давали одинаковые названия) ProDom – белковые домены AsMamDB – альтернативный сплайсинг у млекопитающих
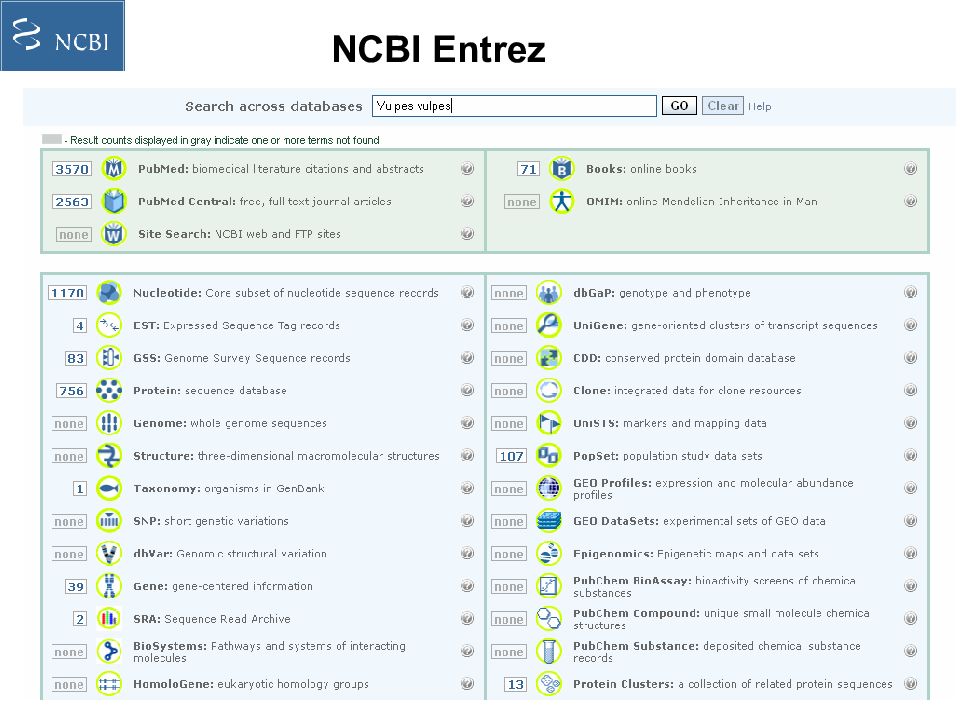
Слайд 12: Интегрированные БД
Вся информация (курируемая, не курируемая) свалена в кучу, и введя имя гена, можно найти всю связанную с ним информацию – в каких организмах встречается, в каком месте генома локализован, какие функции выполняет и т.д. NCBI Entrez – доступ к информации о нуклеотидных и аминокислотных последовательностях и структурах Ecocyc – все о E. coli – гены, белки, метаболизм и пр.
Слайд 13: Локальные БД
DIGAP – база данных аннотированных геномов фитопатогенов PlnTFDB – база данных о транскрипционных факторах растений PRGdb – база данных для анализа генов устойчивости у растений SALAD – база данных для анализа мотивов белков растений и установления их функций SolEST – база данных для изучения транскрипции генов картофеля AnimalTFDB - база данных транскрипционных факторах животных И др.
Слайд 15: GenBank
Аннотированная коллекция всех ДНК сиквенсов Подбазы интегрированы Включает подбазы : GSS – случайные сиквенсы геномной ДНК EST - секвенированные фрагменты экспрессирующихся последовательностей Nucleotide - аннотированные нуклеотидные последовательности И др. Содержит более 10 000 000 сиквенсов растений
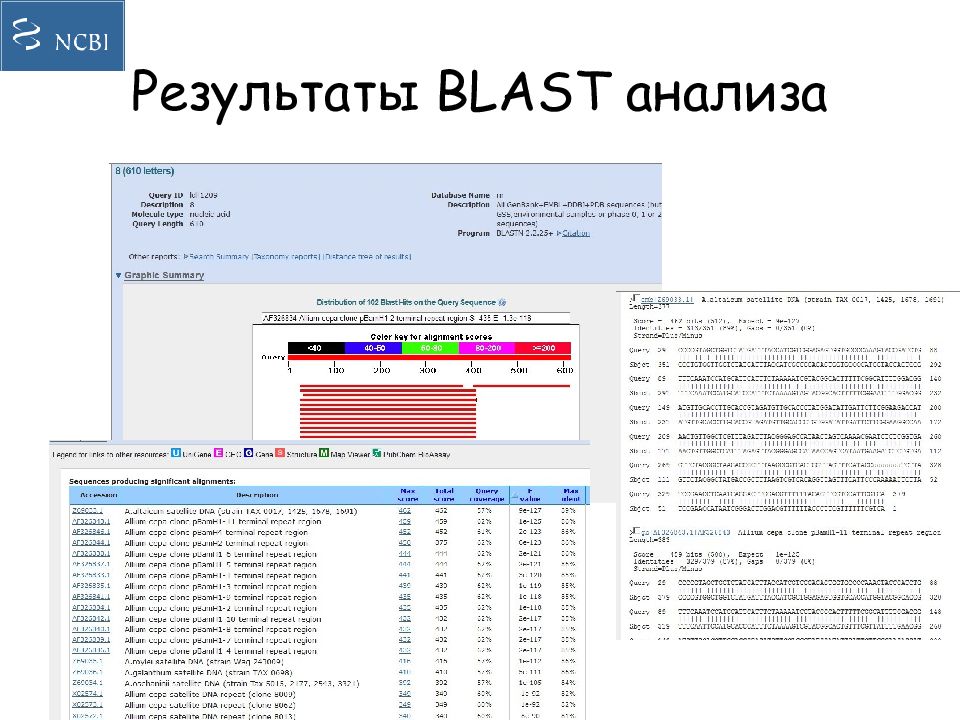
Слайд 18: BLAST (Basic Local Alignment Search Tool
BLASTp (выравнивание аминокислотных последовательностей), BLASTn (выравнивание нуклеотидных последовательностей), BLASTx (выравнивание всех возможных транслятов нашей нуклеотидной последовательности против банка аминокислотных последовательностей), TBLASTx (выравнивание всех возможных транслятов нашей нуклеотидной последовательности против всех транслятов банка нуклеотидных последовательностей).
Слайд 20: Выравнивание ( Alignment)
C равнение ДНК сиквенсов ( С lustalW, ClustalX, Phylipe, MAFFT, BLAST…) Множественное выравнивание – выравнивание нескольких сиквенсов Где это можно сделать online : http://www.ebi.ac.uk/Tools/msa/clustalo/ http://www.genebee.msu.su/services/malign_reduced.html http://www.genome.jp/tools/clustalw/
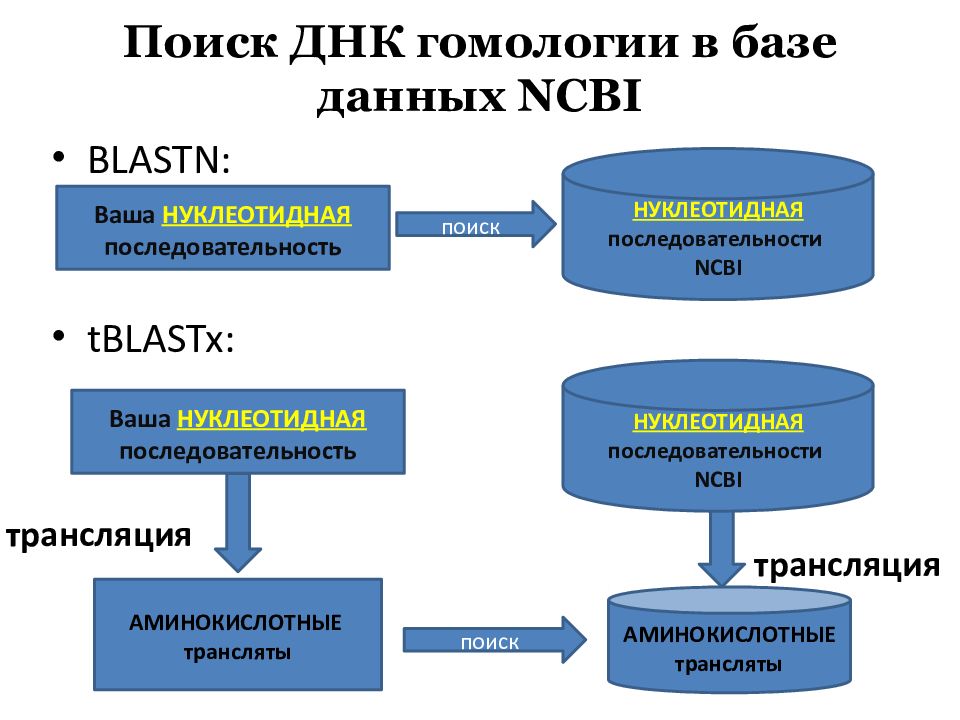
Слайд 21: Поиск ДНК гомологии в базе данных NCBI
BLASTN: tBLASTx: Ваша НУКЛЕОТИДНАЯ последовательность НУКЛЕОТИДНАЯ последовательности NCBI поиск Ваша НУКЛЕОТИДНАЯ последовательность поиск НУКЛЕОТИДНАЯ последовательности NCBI АМИНОКИСЛОТНЫЕ трансляты трансляция АМИНОКИСЛОТНЫЕ трансляты трансляция
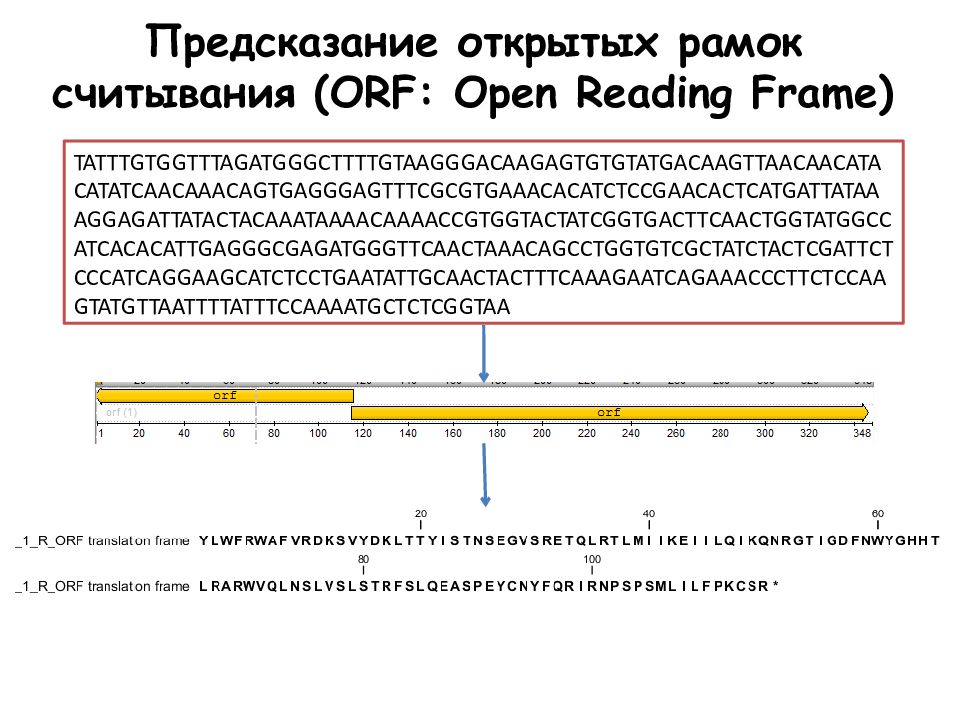
Слайд 24: Предсказание открытых рамок считывания (ORF: Open Reading Frame)
TATTTGTGGTTTAGATGGGCTTTTGTAAGGGACAAGAGTGTGTATGACAAGTTAACAACATACATATCAACAAACAGTGAGGGAGTTTCGCGTGAAACACATCTCCGAACACTCATGATTATAAAGGAGATTATACTACAAATAAAACAAAACCGTGGTACTATCGGTGACTTCAACTGGTATGGCCATCACACATTGAGGGCGAGATGGGTTCAACTAAACAGCCTGGTGTCGCTATCTACTCGATTCTCCCATCAGGAAGCATCTCCTGAATATTGCAACTACTTTCAAAGAATCAGAAACCCTTCTCCAAGTATGTTAATTTTATTTCCAAAATGCTCTCGGTAA
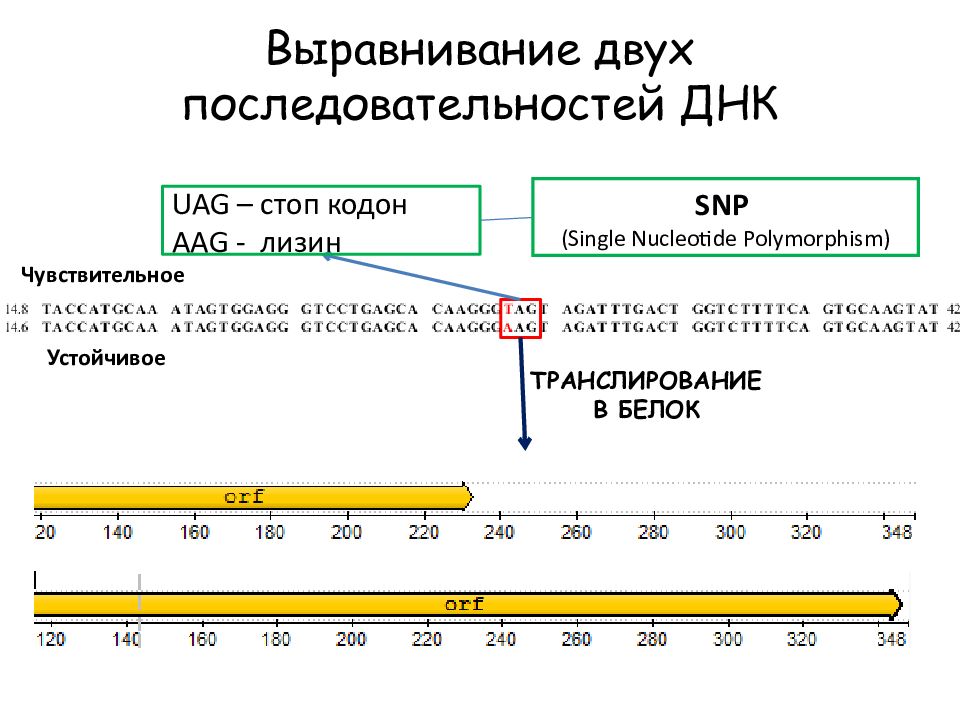
Слайд 26: Выравнивание двух последовательностей ДНК
SNP (Single Nucleotide Polymorphism) UAG – стоп кодон AAG - лизин ТРАНСЛИРОВАНИЕ В БЕЛОК Устойчивое Чувствительное
Слайд 27
Множественное выравнивание ( alignment) ДНК последовательностей с помощью CLC sequence viewer
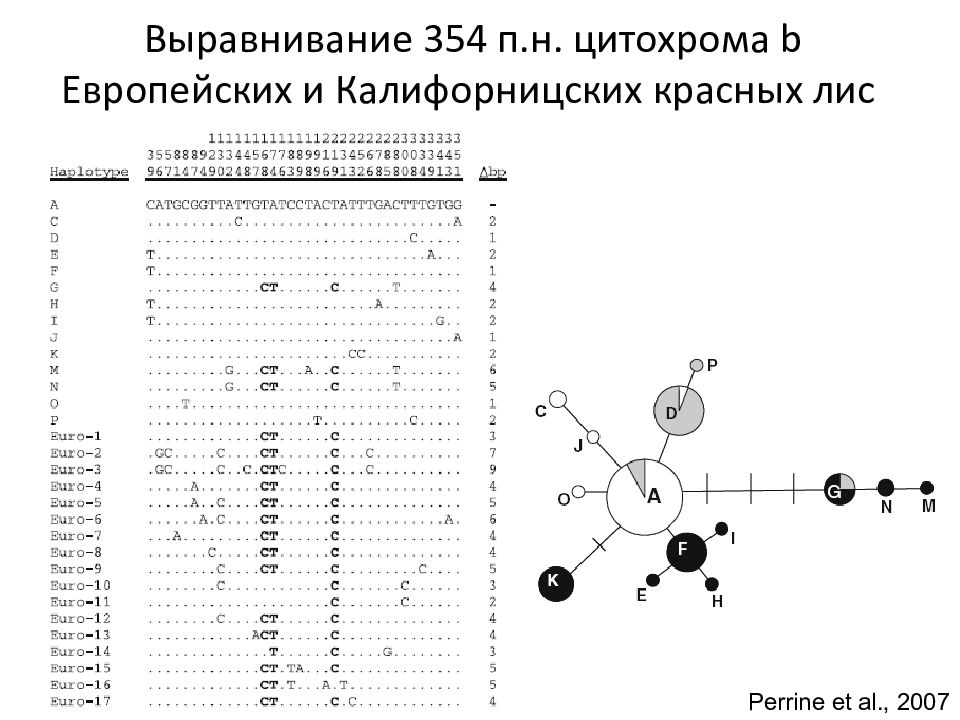
Слайд 28: Выравнивание 354 п.н. цитохрома b Европейских и Калифорницских красных лис
Perrine et al., 2007
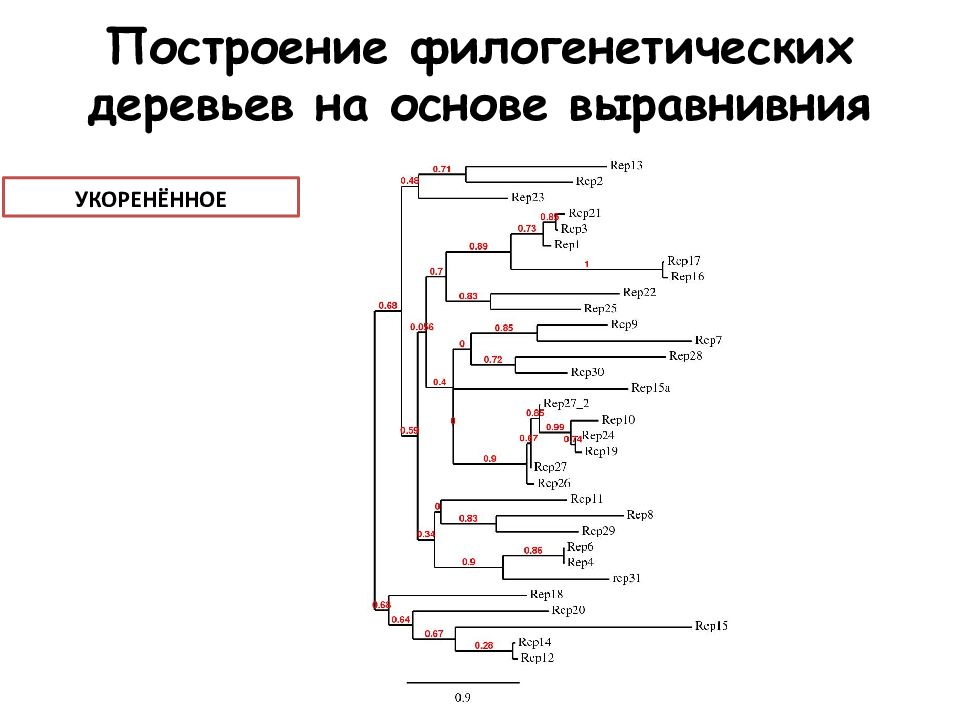
УКОРЕНЁННОЕ Построение филогенетических деревьев на основе выравнивния
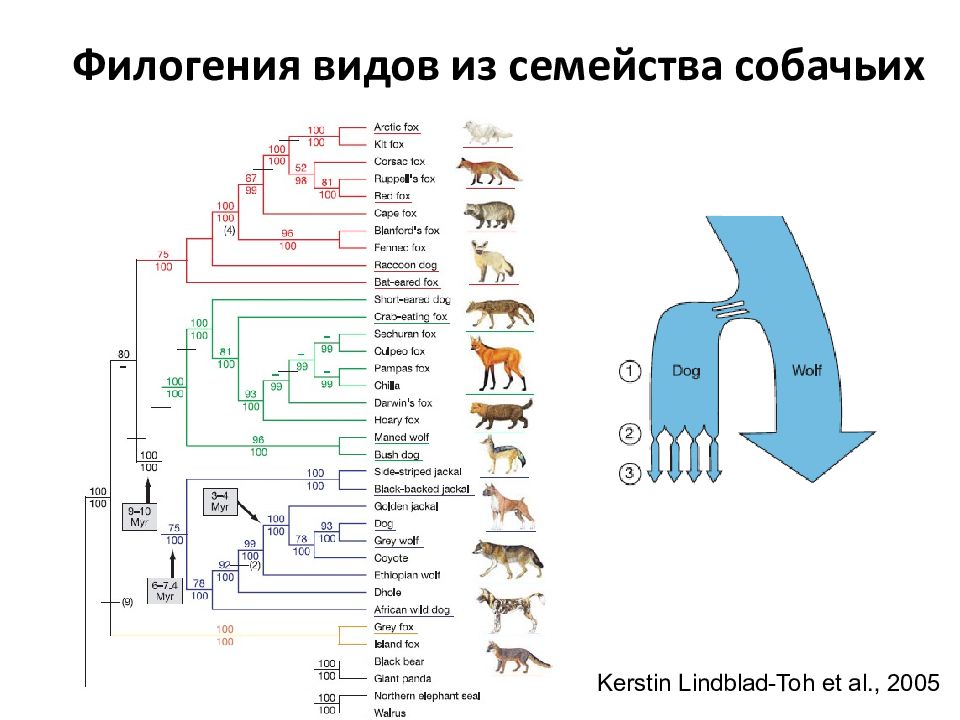
Слайд 30: Филогения видов из семейства собачьих
Kerstin Lindblad-Toh et al., 2005
Слайд 32: SNP – однонуклеотидные изменения
Полиморфизм по одного нуклеотида Встречаются часто Могут возникать в экзонах, интронах и межгенных регионах Могут приводить к изменению в аминокислотной последовательности (не синонимичные) или нет (синонимичные)